Dynamiczna sieć bayesowska - Dynamic Bayesian network
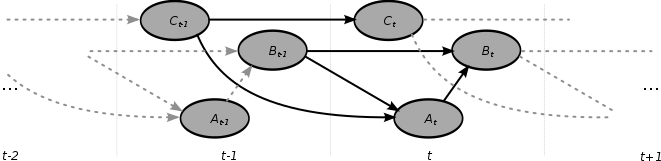
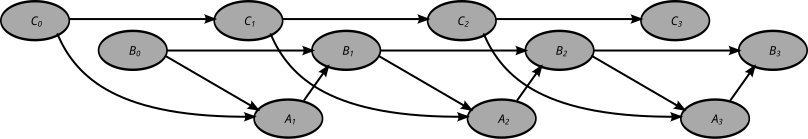
Dynamiczne Bayesa sieci (DBN) jest Bayesa sieci (BN), który odnosi się do zmiennych siebie w sąsiednich przedziałach czasu. Jest to często nazywane BN z dwoma odcinkami czasu (2TBN), ponieważ mówi, że w dowolnym momencie czasu T wartość zmiennej można obliczyć na podstawie wewnętrznych regresorów i bezpośrednio poprzedzającej wartości (czas T-1). DBN zostały opracowane przez Paula Daguma na początku lat 90. w Sekcji Informatyki Medycznej Uniwersytetu Stanforda . Dagum opracował DBN w celu ujednolicenia i rozszerzenia tradycyjnych liniowych modeli w przestrzeni stanów, takich jak filtry Kalmana , liniowych i normalnych modeli prognostycznych, takich jak ARMA oraz prostych modeli zależności, takich jakukryte modele Markowa w ogólną probabilistyczną reprezentację i mechanizm wnioskowania dla dowolnych nieliniowych i nienormalnych dziedzin zależnych od czasu.
Obecnie DBN są powszechne w robotyce i wykazują potencjał dla szerokiej gamy aplikacji do eksploracji danych . Na przykład były wykorzystywane w rozpoznawaniu mowy , kryminalistyce cyfrowej , sekwencjonowaniu białek i bioinformatyce . DBN to uogólnienie ukrytych modeli Markowa i filtrów Kalmana .
DBN są koncepcyjnie powiązane z probabilistycznymi sieciami boolowskimi i podobnie mogą być używane do modelowania systemów dynamicznych w stanie ustalonym.
Zobacz też
Bibliografia
Dalsza lektura
- Murphy, Kevina (2002). Dynamiczne sieci bayesowskie: reprezentacja, wnioskowanie i uczenie się . UC Berkeley, Wydział Informatyki.
- Ghahramani, Zoubin (1997). Nauka dynamicznych sieci bayesowskich . Notatki z wykładów z informatyki . 1387 . s. 168-197. CiteSeerX 10.1.1.56.7874 . doi : 10.1007/BFb0053999 . Numer ISBN 978-3-540-64341-8.
- Friedman, N.; Murphy, K.; Russell, S. (1998). Poznanie budowy dynamicznych sieci probabilistycznych . UAI'98. Morgana Kaufmanna. s. 139–147. CiteSeerX 10.1.1.75.2969 .
Oprogramowanie
- bnt na GitHub : Bayes Net Toolbox for Matlab, autorstwa Kevina Murphy'ego (wydany na licencji GPL )
- Graphical Models Toolkit (GMTK): publicznie dostępny zestaw narzędzi typu open source do szybkiego prototypowania modeli statystycznych przy użyciu dynamicznych modeli graficznych (DGM) i dynamicznych sieci bayesowskich (DBN). GMTK może być używany do zastosowań i badań w zakresie przetwarzania mowy i języka, bioinformatyki, rozpoznawania aktywności i dowolnych aplikacji szeregów czasowych.
- DBmcmc : Inferring Dynamic Bayesian Networks za pomocą MCMC, dla Matlab (bezpłatne oprogramowanie)
- GlobalMIT Matlab toolbox w Google Code : Modelowanie sieci regulacji genów poprzez globalną optymalizację dynamicznej sieci bayesowskiej (wydany na licencji GPL )
- libDAI : biblioteka C++ dostarczająca implementacje różnych (przybliżonych) metod wnioskowania dla dyskretnych modeli graficznych; obsługuje wykresy arbitralnych czynników ze zmiennymi dyskretnymi, w tym dyskretnymi polami losowymi Markowa i sieciami Bayesian (wydanymi na licencji FreeBSD )
- aGrUM : biblioteka C++ (z wiązaniami Pythona) dla różnych typów PGM, w tym Bayesian Networks i Dynamic Bayesian Networks (wydana na licencji GPLv3)
- FALCON : Zestaw narzędzi Matlab do kontekstualizacji modeli DBN sieci regulacyjnych z biologicznymi danymi ilościowymi, w tym różne schematy regularyzacji w celu modelowania wcześniejszej wiedzy biologicznej (wydany na licencji GPLv3)