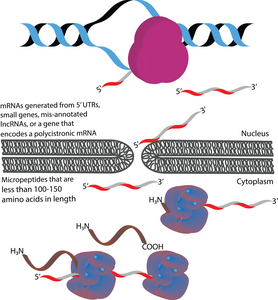
Mikropeptyd - Micropeptide
Mikropeptydy (określane również jako mikroproteiny) to polipeptydy o długości mniejszej niż 100-150 aminokwasów, które są kodowane przez krótkie otwarte ramki odczytu (sORF). Pod tym względem różnią się one od wielu innych aktywnych małych polipeptydów, które są wytwarzane przez potranslacyjne cięcie większych polipeptydów. Pod względem wielkości mikropeptydy są znacznie krótsze niż białka „kanoniczne”, które mają średnią długość 330 i 449 aminokwasów odpowiednio u prokariontów i eukariontów. Mikropeptydy są czasami nazywane zgodnie z ich lokalizacją w genomie. Na przykład, translacji produkt upstream otwartej ramki odczytu (uORF) można nazwać peptydem kodowanym przez uORF (uPEP). Mikropeptydy nie posiadają N-końcowych sekwencji sygnałowych, co sugeruje, że prawdopodobnie są zlokalizowane w cytoplazmie . Jednak niektóre mikropeptydy znaleziono w innych przedziałach komórkowych, na co wskazuje istnienie mikropeptydów transbłonowych . Występują zarówno u prokariontów, jak i eukariotów. sORF, z których ulegają translacji mikropeptydy, mogą być kodowane w 5' UTR , małych genach lub policistronowych mRNA . Niektóre kodujące mikropeptydy geny zostały pierwotnie błędnie opisane jako długie niekodujące RNA (lncRNA).
Biorąc pod uwagę ich mały rozmiar, sORF były początkowo pomijane. Jednak setki tysięcy domniemanych mikropeptydów zidentyfikowano różnymi technikami w wielu organizmach. Tylko niewielka część tych z potencjałem kodowania potwierdzono ich ekspresję i funkcję. Te, które zostały scharakteryzowane funkcjonalnie, ogólnie odgrywają role w sygnalizacji komórkowej , organogenezie i fizjologii komórkowej . Im więcej mikropeptydów zostaje odkrytych, tym więcej ich funkcji. Jedną z funkcji regulacyjnych jest funkcja peptoprzełączników, które hamują ekspresję dalszych sekwencji kodujących poprzez blokowanie rybosomów , poprzez ich bezpośrednią lub pośrednią aktywację przez małe cząsteczki.
Identyfikacja
Istnieją różne techniki eksperymentalne identyfikacji potencjalnych ORF i ich produktów translacyjnych. Techniki te są przydatne jedynie do identyfikacji sORF, które mogą wytwarzać mikropeptydy, a nie do bezpośredniej charakterystyki funkcjonalnej.
Sekwencjonowanie RNA
Jedną z metod znajdowania potencjalnych sORF, a zatem mikropeptydów, jest sekwencjonowanie RNA ( RNA-Seq ). RNA-Seq wykorzystuje sekwencjonowanie nowej generacji (NGS) w celu określenia, które RNA ulegają ekspresji w danej komórce, tkance lub organizmie w określonym momencie. Ten zbiór danych, znany jako transkryptom , może być następnie wykorzystany jako źródło do znajdowania potencjalnych sORF. Ze względu na duże prawdopodobieństwo przypadkowego wystąpienia sORF mniejszych niż 100 aa, konieczne są dalsze badania w celu określenia prawdziwości danych uzyskanych tą metodą.
Profilowanie rybosomów (Ribo-Seq)
Profilowanie rybosomów zostało wykorzystane do identyfikacji potencjalnych mikropeptydów w coraz większej liczbie organizmów, w tym muszek owocowych, danio pręgowanego, myszy i ludzi. Jedna metoda wykorzystuje związki takie jak harringtonina, puromycyna lub laktymidomycyna do zatrzymania rybosomów w miejscach inicjacji translacji. Wskazuje, gdzie odbywa się aktywne tłumaczenie. Inhibitory wydłużania translacji, takie jak emetyna lub cykloheksymid, mogą być również stosowane do uzyskania śladów rybosomów, które z większym prawdopodobieństwem prowadzą do translacji ORF. Jeśli rybosom jest związany na lub w pobliżu sORF, przypuszczalnie koduje mikropeptyd.
Spekrtometria masy
Spektrometria mas (MS) to złoty standard identyfikacji i sekwencjonowania białek. Stosując tę technikę, badacze są w stanie określić, czy polipeptydy faktycznie ulegają translacji z sORF.
Zastosowania proteogenomiczne
Proteogenomika łączy proteomikę, genomikę i transcyptomikę. Jest to ważne przy poszukiwaniu potencjalnych mikropeptydów. Jedna metoda wykorzystania proteogenomiki obejmuje wykorzystanie danych RNA-Seq do stworzenia niestandardowej bazy danych wszystkich możliwych polipeptydów. Przeprowadza się chromatografię cieczową, a następnie tandem MS (LC-MS/MS) w celu dostarczenia informacji o sekwencji produktów translacji. Porównanie danych transkryptomicznych i proteomicznych można wykorzystać do potwierdzenia obecności mikropeptydów.
Ochrona filogenetyczna
Konserwacja filogenetyczna może być użytecznym narzędziem, szczególnie podczas przeszukiwania dużej bazy danych sORF. Prawdopodobieństwo powstania funkcjonalnego mikropeptydu przez sORF jest bardziej prawdopodobne, jeśli jest on konserwowany w wielu gatunkach. Jednak nie będzie to działać dla wszystkich sORF. Na przykład te, które są kodowane przez lncRNA, mają mniejsze prawdopodobieństwo, że zostaną zachowane, biorąc pod uwagę, że same lncRNA nie mają wysokiej konserwacji sekwencji. Konieczne będą dalsze eksperymenty w celu ustalenia, czy faktycznie wytwarzany jest funkcjonalny mikropeptyd.
Walidacja potencjału kodowania białek
Przeciwciała
Niestandardowe przeciwciała ukierunkowane na interesujący mikropeptyd mogą być przydatne do ilościowego oznaczania ekspresji lub określania lokalizacji wewnątrzkomórkowej. Jak w przypadku większości białek, niska ekspresja może utrudnić wykrywanie. Mały rozmiar mikropeptydu może również prowadzić do trudności w zaprojektowaniu epitopu, z którego ma być skierowane przeciwciało.
Tagowanie za pomocą CRISPR-Cas9
Edycja genomu może być wykorzystana do dodania FLAG/MYC lub innych małych znaczników peptydowych do endogennego sORF, tworząc w ten sposób białka fuzyjne. W większości przypadków ta metoda jest korzystna, ponieważ można ją wykonać szybciej niż opracowanie niestandardowego przeciwciała. Jest również użyteczny w przypadku mikropeptydów, dla których nie można nakierować epitopu.
Tłumaczenie in vitro
Proces ten obejmuje klonowanie pełnej długości cDNA mikropeptydu do plazmidu zawierającego promotor T7 lub SP6. Metoda ta wykorzystuje bezkomórkowy system syntezy białek w obecności 35S -metioniny do wytworzenia peptydu będącego przedmiotem zainteresowania. Produkty można następnie analizować za pomocą elektroforezy żelowej, a peptyd znakowany 35S wizualizuje się za pomocą autoradiografii.
Bazy danych i repozytoria
Istnieje kilka repozytoriów i baz danych, które zostały utworzone zarówno dla sORF, jak i mikropeptydów. Repozytorium małych ORF odkrytych przez profilowanie rybosomów można znaleźć na sORFs.org. Repozytorium przypuszczalnych peptydów kodowanych przez sORF w Arabidopsis thaliana można znaleźć w ARA-PEP. Bazę danych małych białek, zwłaszcza kodowanych przez niekodujące RNA, można znaleźć w SmProt.
Przykłady prokariotyczne
Do tej pory większość mikropeptydów zidentyfikowano w organizmach prokariotycznych . Chociaż większość z tych, które zostały zbadane, nie została jeszcze w pełni scharakteryzowana, wiele wydaje się mieć kluczowe znaczenie dla przetrwania tych organizmów. Ze względu na swój niewielki rozmiar prokariota są szczególnie podatne na zmiany w swoim środowisku i jako takie wypracowały metody zapewniające ich egzystencję.
Escherichia coli (E. coli)
Mikropeptydy eksprymowane w E. coli stanowią przykład bakteryjnych adaptacji środowiskowych. Większość z nich podzielono na trzy grupy: peptydy liderowe, białka rybosomalne i białka toksyczne. Białka liderowe regulują transkrypcję i/lub translację białek biorących udział w metabolizmie aminokwasów, gdy aminokwasów jest mało. Białka rybosomalne obejmują L36 ( rpmJ ) i L34 ( rpmH ), dwa składniki podjednostki rybosomalnej 50S. Toksyczne białka, takie jak ldrD , są toksyczne na wysokim poziomie i mogą zabijać komórki lub hamować wzrost, co zmniejsza żywotność komórki gospodarza.
Salmonella enterica (S. enterica)
W S. enterica czynnik wirulencji MgtC bierze udział w adaptacji do środowisk o niskiej zawartości magnezu. Hydrofobowy peptyd MgrR wiąże się z MgtC, powodując jego degradację przez proteazę FtsH.
Bacillus subtilis (B. subtilis)
Mikropeptyd 46 aminokwasów Sda, wyrażany przez B. subtilis , hamuje sporulacji, gdy inicjacja replikacji jest osłabiona. Hamując kinazę histydynową KinA, Sda zapobiega aktywacji czynnika transkrypcyjnego Spo0A, który jest niezbędny do sporulacji.
Staphylococcus aureus (S. aureus)
W S. aureus występuje grupa mikropeptydów 20-22 aminokwasów, które są wydalane podczas infekcji gospodarza w celu przerwania błon neutrofilowych, powodując lizę komórek. Te mikropeptydy pozwalają bakterii uniknąć degradacji przez główne mechanizmy obronne ludzkiego układu odpornościowego.
Przykłady eukariotyczne
Mikropeptydy odkryto w organizmach eukariotycznych od Arabidopsis thaliana po ludzi. Odgrywają różne role w rozwoju tkanek i narządów, a także w utrzymaniu i funkcjonowaniu po pełnym rozwinięciu. Chociaż wiele z nich nie zostało jeszcze funkcjonalnie scharakteryzowanych, a prawdopodobnie więcej pozostaje do odkrycia, poniżej znajduje się podsumowanie ostatnio zidentyfikowanych funkcji mikropeptydów eukariotycznych.
Arabidopsis thaliana ( A. thaliana )
Polaris (PLS) koduje micropeptide 36 aa. Jest niezbędny do prawidłowego ułożenia naczyniowego liści i ekspansji komórek w korzeniu. Ten mikropeptyd oddziałuje z rozwojowymi białkami PIN, tworząc sieć krytyczną dla przenikania hormonalnego między auksyną, etylenem i cytokininą.
ROTUNDIFOLIA (ROT4 ) w A. thaliana koduje peptyd o długości 53 aminokwasów, który lokalizuje się w błonie komórkowej komórek liściowych. Mechanizm działania ROT4 nie jest dobrze poznany, ale mutanty mają krótkie zaokrąglone liście, co wskazuje, że ten peptyd może być ważny w morfogenezie liści.
Zea mays ( Z. mays )
Brick1 (Brk1) koduje mikropeptyd 76 aminokwasów, który jest wysoce konserwatywny zarówno u roślin, jak iu zwierząt. Stwierdzono , że u Z. mays bierze udział w morfogenezie nabłonka liścia poprzez promowanie wielu zależnych od aktyny zdarzeń polaryzacji komórek w rozwijającym się naskórku liścia. Zm401p10 jest mikropeptydem 89 aa, który odgrywa rolę w prawidłowym rozwoju pyłku w tapetum. Po mitozie ma również zasadnicze znaczenie w degradacji tapetum. Zm908p11 to mikropeptyd o długości 97 aminokwasów, kodowany przez gen Zm908, który ulega ekspresji w dojrzałych ziarnach pyłku. Lokalizuje się w cytoplazmie łagiewek pyłkowych, gdzie wspomaga ich wzrost i rozwój.
Drosophila melanogaster ( D. melanogaster )
Ewolucyjnie konserwowany gen polerowanego ryżu ( pri ) u D. melanogaster , znany jako tarsal-less (tal) jest zaangażowany w różnicowanie naskórka. Ten policistronowy transkrypt koduje cztery podobne peptydy o długości od 11 do 32 aminokwasów. Funkcjonują w celu obcięcia czynnika transkrypcyjnego Shavenbaby (Svb). Przekształca to Svb w aktywator, który bezpośrednio reguluje ekspresję docelowych efektorów, w tym miniaturowych (m) i shavenoidów (sha) , które razem odpowiadają za tworzenie trichomów.
Danio rerio ( D. rerio )
Elabela gen ( Ela) (aka Apela, maluch) jest ważna dla embriogenezy. Jest szczególnie wyrażany w późnych stadiach blastuli i gastruli. Podczas gastrulacji ma kluczowe znaczenie w promowaniu internalizacji i ukierunkowanych na zwierzę-biegunach ruchu komórek mez- endodermy . Po gastrulacji Ela jest wyrażana w bocznej mezodermie, endodermie, a także w przedniej i tylnej strunie grzbietowej. Chociaż został oznaczony jako lncRNA u danio pręgowanego , myszy i człowieka, stwierdzono, że 58-aa ORF jest wysoce konserwatywny wśród gatunków kręgowców. Ela jest przetwarzana przez usunięcie jej N-końcowego peptydu sygnałowego, a następnie wydzielana do przestrzeni zewnątrzkomórkowej . Jego 34-aa dojrzały peptyd służy jako pierwszy endogenny ligand dla GPCR znanego jako receptor apelinowy . Inaktywacji genetycznej Ela lub Aplnr w danio skutkuje fenotypem bez serca.
Mus musculus ( M. musculus )
Mioregulina ( Mln ) jest kodowana przez gen pierwotnie oznaczony jako lncRNA. Mln ulega ekspresji we wszystkich 3 rodzajów mięśni szkieletowych i działa podobnie do mikropeptydów fosfolamban (PLN) w mięśniu sercowym i sarcolipin (SLN) w zwolnionym (typ I), mięśniach szkieletowych. Te mikropeptydów interakcję z sarkoplazmatycznej Ca 2+ -ATPazy (serca), pompę membranową odpowiedzialnego za regulację Ca 2+ wychwyt do sarkoplazmatycznej (SR). Hamując wychwyt Ca 2+ do SR, powodują rozluźnienie mięśni. Podobnie, geny endoreguliny (ELN) i innej reguliny (ALN) kodują mikropeptydy transbłonowe, które zawierają motyw wiążący SERCA i są konserwatywne u ssaków.
Myomixer (Mymx) jest kodowany przez gen Gm7325, specyficzny dla mięśni peptyd o długości 84 aminokwasów, który odgrywa rolę podczas embriogenezy w fuzji i tworzeniu mięśni szkieletowych. Lokalizuje się na błonie komórkowej, kojarząc się z fuzogennym białkiem błonowym Myomaker (Mymk). U ludzi gen kodujący Mymx jest oznaczony jako niescharakteryzowany LOC101929726 . Ortologi znajdują się również w genomach żółwi, żab i ryb.
Homo sapiens ( H. sapiens )
U ludzi NoBody (polipeptyd dysocjujący ciała P bez adnotacji), mikropeptyd o długości 68 aminokwasów, został odkryty w długim niekodującym RNA (lincRNA) LINC01420 . Ma wysoką konserwację sekwencji wśród ssaków i lokalizuje się w P-ciałach . Wzbogaca białka związane z dekapowaniem 5' mRNA . Uważa się, że oddziałuje bezpośrednio z Enhancer of mRNA Decapping 4 (EDC4).
ELABELA ( ELA) (aka APELA) to endogenny hormon wydzielany jako 32-aminokwasowy mikropeptyd przez ludzkie embrionalne komórki macierzyste . Niezbędne jest utrzymanie samoodnowy i pluripotencji ludzkich embrionalnych komórek macierzystych . Jego sygnały w sposób autokrynny poprzezszlak PI3/AKT przez niezidentyfikowany receptor powierzchniowy komórki . W różnicowaniu komórek mezoendermalnych ELA wiąże się i sygnalizuje poprzez APLNR , GPCR, który może również reagować na peptyd hormonalny APLN .
Przewiduje się, że gen C7orf49 , zachowany u ssaków, po alternatywnym splicingu wytwarza trzy mikropeptydy. Wcześniej stwierdzono, że MRI-1 jest modulatorem infekcji retrowirusem. Drugi przewidywany mikropeptyd, MRI-2, może być ważny w łączeniu niehomologicznych końców (NHEJ) pęknięć dwuniciowych DNA. W eksperymentach z ko-immunoprecypitacją, MRI-2 wiązał się z Ku70 i Ku80 , dwiema podjednostkami Ku , które odgrywają główną rolę w szlaku NHEJ.
Mikropeptyd składający się z 24 aminokwasów, humanina (HN), oddziałuje z białkiem X (Bax) wywołującym apoptozę . W stanie aktywnym Bax przechodzi zmianę konformacyjną, która odsłania domeny celujące w błonę. Powoduje to, że przemieszcza się z cytozolu do błony mitochondrialnej, gdzie wstawia i uwalnia białka apoptogenne, takie jak cytochrom c. Poprzez interakcję z Bax, HN zapobiega celowaniu Bax w mitochondria, blokując w ten sposób apoptozę.
Stwierdzono, że mikropeptyd 90aa, " Mały polipeptyd regulacyjny odpowiedzi aminokwasowej " lub SPAAR, jest kodowany w lncRNA LINC00961 . Jest konserwowany między człowiekiem a myszą i lokalizuje się w późnym endosomie/lizosomie. SPAAR oddziałuje z czterema podjednostkami kompleksu v-ATPazy , hamując translokację mTORC1 na powierzchnię lizosomów, gdzie jest aktywowany. Regulacja w dół tego mikropeptydu umożliwia aktywację mTORC1 poprzez stymulację aminokwasów, promując regenerację mięśni.
Bibliografia
![]() Ten artykuł został zaadaptowany z następującego źródła na licencji CC BY 4.0 ( 2018 ) ( raporty recenzentów ):
Maria E. Sousa; Michael H. Farkas (13 grudnia 2018). „Mikropeptyd” . PLOS Genetyka . 14 (12): e1007764. doi : 10.1371/JOURNAL.PGEN.1007764 . ISSN 1553-7390 . PMC 6292567 . PMID 30543625 . Wikidane Q60017699 .
Ten artykuł został zaadaptowany z następującego źródła na licencji CC BY 4.0 ( 2018 ) ( raporty recenzentów ):
Maria E. Sousa; Michael H. Farkas (13 grudnia 2018). „Mikropeptyd” . PLOS Genetyka . 14 (12): e1007764. doi : 10.1371/JOURNAL.PGEN.1007764 . ISSN 1553-7390 . PMC 6292567 . PMID 30543625 . Wikidane Q60017699 .
- ^ B c d e f g Crappé J, Van Criekinge W, Menschaert G (2014). „Małe rzeczy sprawiają, że dzieją się wielkie rzeczy: podsumowanie genów kodujących mikropeptydy” . Otwarta proteomika EuPA . 3 : 128–137. doi : 10.1016/j.euprot.2014.02.006 .
- ^ B c d e f g Makarewich CA Olson PL (wrzesień 2017). „Wydobywanie mikropeptydów” . Trendy w biologii komórki . 27 (9): 685–696. doi : 10.1016/j.tcb.2017.04.006 . PMC 5565689 . PMID 28528987 .
- ^ Guillén G, Diaz-Camino C, Loyola-Torres CA, Aparicio-Fabre R, Hernández-López A, Diaz-Sánchez M, Sanchez F (2013). „Szczegółowa analiza domniemanych genów kodujących małe białka w genomach roślin strączkowych” . Granice w naukach o roślinach . 4 : 208. doi : 10.3389/fpls.2013.00208 . PMC 3687714 . PMID 23802007 .
- ^ Hashimoto Y, Kondo T, Kageyama Y (czerwiec 2008). „Liliputi stają w centrum uwagi: nowa klasa małych genów peptydowych w morfogenezie” . Rozwój, wzrost i zróżnicowanie . 50 Suplement 1: S269–76. doi : 10.1111/j.1440-169x.2008.0994.x . PMID 18459982 .
- ^ Zhang J (marzec 2000). „Rozkłady długości białka dla trzech domen życia”. Trendy w genetyce . 16 (3): 107-9. doi : 10.1016/s0168-9525(99)01922-8 . PMID 10689349 .
- ^ Rothnagel J, Menschaert G (maj 2018). „Krótkie otwarte ramki odczytu i ich zakodowane peptydy” . Proteomika . 18 (10): e1700035. doi : 10.1002/pmic.201700035 . PMID 29691985 .
- ^ a b Anderson DM, Anderson KM, Chang CL, Makarewich CA, Nelson BR, McAnally JR, Kasaragod P, Shelton JM, Liou J, Bassel-Duby R, Olson EN (luty 2015). „Mikropeptyd kodowany przez przypuszczalnie długi niekodujący RNA reguluje wydajność mięśni” . Komórka . 160 (4): 595-606. doi : 10.1016/j.cell.2015.01.09 . PMC 4356254 . PMID 25640239 .
- ^ B Bi P Ramirez-Martinez A, Li H, Cannavino J McAnally JR Shelton JM, Sanchez-Ortiz E Basell-Duby R Olson PL (kwiecień 2017). „Kontrola tworzenia mięśni przez fuzogenny myomixer mikropeptydów” . Nauka . 356 (6335): 323-327. Kod Bibcode : 2017Sci...356..323B . doi : 10.1126/science.aam9361 . PMC 5502127 . PMID 28386024 .
- ^ B Alix E Blanc-Potard AB (luty 2008). „Wspomagana peptydowo degradacja czynnika wirulencji Salmonella MgtC” . Dziennik EMBO . 27 (3): 546–57. doi : 10.1038/sj.emboj.7601983 . PMC 2241655 . PMID 18200043 .
- ^ B Burkholder WF Kurtser I Grossman AD (styczeń 2001). „Białka inicjacji replikacji regulują rozwojowy punkt kontrolny w Bacillus subtilis”. Komórka . 104 (2): 269–79. doi : 10.1016/s0092-8674(01)00211-2 . hdl : 1721.1/83916 . PMID 11207367 . S2CID 15048130 .
- ^ B c d e Andrews SJ Rothnagel JA (marzec 2014). „Pojawiające się dowody funkcjonalnych peptydów kodowanych przez krótkie otwarte ramki odczytu”. Recenzje przyrody. Genetyka . 15 (3): 193-204. doi : 10.1038/nrg3520 . PMID 24514441 . S2CID 22543778 .
- ^ a b Bazzini AA, Johnstone TG, Christiano R, Mackowiak SD, Obermayer B, Fleming ES, Vejnar CE, Lee MT, Rajewsky N, Walther TC, Giraldez AJ (maj 2014). „Identyfikacja małych ORF u kręgowców za pomocą śladów rybosomów i ochrony ewolucyjnej” . Dziennik EMBO . 33 (9): 981-93. doi : 10.1002/embj.201488411 . PMC 4193932 . PMID 24705786 .
- ^ Ingolia NT, Brar GA, Stern-Ginossar N, Harris MS, Talhouarne GJ, Jackson SE, Wills MR, Weissman JS (wrzesień 2014). „Profilowanie rybosomów ujawnia wszechobecną translację poza opisanymi genami kodującymi białka” . Raporty komórkowe . 8 (5): 1365–79. doi : 10.1016/j.celrep.2014.07.045 . PMC 4216110 . PMID 25159147 .
- ^ Stern-Ginossar N, Ingolia NT (listopad 2015). „Profilowanie rybosomów jako narzędzie do rozszyfrowania złożoności wirusów”. Roczny przegląd wirusologii . 2 (1): 335–49. doi : 10.1146/annurev- virology-100114-054854 . PMID 26958919 .
- ^ „sORFs.org: repozytorium małych ORF zidentyfikowanych przez profilowanie rybosomów” . sorfs.org . Pobrano 2018-12-14 .
- ^ Olexiouk V Crappé J Verbruggen S Verhegen K Martens L Menschaert G (styczeń 2016). „sORFs.org: repozytorium małych ORF zidentyfikowanych przez profilowanie rybosomów” . Badania kwasów nukleinowych . 44 (D1): D324–9. doi : 10.1093/nar/gkv1175 . PMC 4702841 . PMID 26527729 .
- ^ „ARA-PEPs: Repozytorium przypuszczalnych peptydów kodowanych przez sORF w Arabidopsis thaliana ” . www.biw.kuleuven.be . Pobrano 2018-12-14 .
- ^ Hazarika RR De Coninck B Yamamoto LR Martin LR Cammue BP van Noort V (styczeń 2017). „ARA-PEPs: repozytorium domniemanych peptydów kodowanych przez sORF w Arabidopsis thaliana” . Bioinformatyka BMC . 18 (1): 37. doi : 10.1186/s12859-016-1458-y . PMC 5240266 . PMID 28095775 .
- ^ „SmProt: baza danych małych białek zakodowanych przez adnotacje kodujące i niekodujące loci RNA” . bioinfo.ibp.ac.cn . Pobrano 2018-12-14 .
- ^ Hao Y, Zhang L, Niu Y, Cai T, Luo J, He S, Zhang B, Zhang D, Qin Y, Yang F, Chen R (lipiec 2018). „SmProt: baza danych małych białek zakodowanych przez adnotacje kodujące i niekodujące loci RNA”. Odprawy w Bioinformatyce . 19 (4): 636–643. doi : 10.1093/bib/bbx005 . PMID 28137767 .
- ^ Hemm MR Paul BJ Schneider TD Storz G Rudd KE (grudzień 2008). „Małe białka błonowe znalezione w genomice porównawczej i modelach miejsca wiązania rybosomów” . Mikrobiologia molekularna . 70 (6): 1487-501. doi : 10.1111/j.1365-2958.2008.06495.x . PMC 2614699 . PMID 19121005 .
- ^ Wang R Braughton KR Kretschmer D Bach TH Queck SY Li M Kennedy AD Dorward DW Klebanoff SJ Peschel A DeLeo FR Otto M (grudzień 2007). „Identyfikacja nowych peptydów cytolitycznych jako kluczowych wyznaczników wirulencji dla MRSA związanych ze społecznością”. Medycyna przyrodnicza . 13 (12): 1510–4. doi : 10.1038/nm1656 . PMID 17994102 . S2CID 8465052 .
- ^ Hemm MR, Paul BJ, Miranda-Ríos J, Zhang A, Soltanzad N, Storz G (styczeń 2010). „Małe białka odpowiedzi na stres w Escherichia coli: białka pominięte w klasycznych badaniach proteomicznych” . Czasopismo Bakteriologii . 192 (1): 46–58. doi : 10.1128/jb.00872-09 . PMC 2798279 . PMID 19734316 .
- ^ Casson SA, Chilley PM, Topping JF, Evans IM, Souter MA, Lindsey K (sierpień 2002). „Gen POLARIS Arabidopsis koduje przewidywany peptyd niezbędny do prawidłowego wzrostu korzeni i układu naczyniowego liści” . Komórka Roślinna . 14 (8): 1705–21. doi : 10.1105/tpc.002618 . PMC 151460 . PMID 12172017 .
- ^ Chilley PM Casson SA Tarkowski P Hawkins N Wang KL Hussey PJ Beale M Ecker JR Sandberg GK Lindsey K (listopad 2006). „Peptyd POLARIS Arabidopsis reguluje transport auksyny i wzrost korzeni poprzez wpływ na sygnalizację etylenową” . Komórka Roślinna . 18 (11): 3058–72. doi : 10.1105/tpc.106.040790 . PMC 1693943 . PMID 17138700 .
- ^ Liu J, Mehdi S, dolewanie J, Friml J, Lindsey K (2013). "Współdziałanie PLS i PIN oraz przesłuch hormonalny w rozwoju korzeni Arabidopsis" . Granice w naukach o roślinach . 4 : 75. doi : 10.3389/fpls.2013.00075 . PMC 3617403 . PMID 23577016 .
- ^ Narita NN Moore S Horiguchi G Kubo M Demura T Fukuda H Goodrich J Tsukaya H (maj 2004). „Nadekspresja nowego małego peptydu ROTUNDIFOLIA4 zmniejsza proliferację komórek i zmienia kształt liścia u Arabidopsis thaliana”. Dziennik Roślin . 38 (4): 699–713. doi : 10.1111/j.1365-313x.2004.02078.x . PMID 15125775 .
- ^ Frank MJ, Smith LG (maj 2002). „Małe, nowe białko wysoce konserwowane w roślinach i zwierzętach sprzyja spolaryzowanemu wzrostowi i podziałowi komórek naskórka liścia kukurydzy” . Aktualna Biologia . 12 (10): 849–53. doi : 10.1016/s0960-9822(02)00819-9 . PMID 12015123 . S2CID 14725039 .
- ^ Wang D, Li C, Zhao Q, Zhao L, Wang M, Zhu D, Ao G, Yu J (2009). „Zm401p10, kodowany przez gen specyficzny dla pylników z krótkimi otwartymi ramkami odczytu, jest niezbędny do degeneracji tapetum i rozwoju pylników u kukurydzy”. Funkcjonalna Biologia Roślin . 36 (1): 73–85. doi : 10.1071/fp08154 . PMID 32688629 .
- ^ Dong X, Wang D, Liu P, Li C, Zhao Q, Zhu D, Yu J (maj 2013). „Zm908p11, kodowany przez gen krótkiej otwartej ramki odczytu (sORF), działa we wzroście łagiewki pyłkowej jako ligand profiliny w kukurydzy” . Dziennik Botaniki Eksperymentalnej . 64 (8): 2359–72. doi : 10.1093/jxb/ert093 . PMC 3654424 . PMID 23676884 .
- ^ Kondo T Plaza S Zanet J Benrabah E Valenti P Hashimoto Y Kobayashi S Payre F Kageyama Y (lipiec 2010). „Małe peptydy przełączają aktywność transkrypcyjną Shavenbaby podczas embriogenezy Drosophila”. Nauka . 329 (5989): 336-9. Kod Bibcode : 2010Sci...329..336K . doi : 10.1126/science.1188158 . PMID 20647469 . S2CID 2927777 .
- ^ B c Odcięta SC, Ho L, J, Tian REVERSADE B (grudzień 2013). "ELABELA: hormon niezbędny do przekazywania sygnałów rozwojowych serca poprzez receptor apelinowy" . Komórka Rozwojowa . 27 (6): 672–80. doi : 10.1016/j.devcel.2013.11.002 . PMID 24316148 .
- ^ Pauli A, Norris ML, Valen E, Chew GL, Gagnon JA, Zimmerman S, Mitchell A, Ma J, Dubrulle J, Reyon D, Tsai SQ, Joung JK, Saghatelian A, Schier AF (luty 2014). „Maluch: sygnał embrionalny, który promuje ruch komórek za pośrednictwem receptorów Apelin” . Nauka . 343 (6172): 1248636. doi : 10.1126/science.1248636 . PMC 4107353 . PMID 24407481 .
- ^ Deshwar, Ashish R; Chng, spokojny C; Ho, Lena; Rewers, Bruno; Scott, Ian C (14.04.2016). Robertson, Elżbieta (red.). „Receptor Apelin wzmacnia sygnalizację Nodal/TGFβ w celu zapewnienia prawidłowego rozwoju serca” . e-życie . 5 : e13758. doi : 10.7554/eLife.13758 . ISSN 2050-084X . PMC 4859801 . PMID 27077952 .
- ^ Scott, Ian C.; Masriego, Bernarda; D'Amico, Leonard A.; Jin, Suk-Won; Jungblut, Benno; Wehman, Ann M.; Baier, Herwig; Audigier, Yves; Stainier, Didier YR (marzec 2007). „Receptor agtrl1b sprzężony z białkiem g reguluje wczesny rozwój progenitorów mięśnia sercowego” . Komórka Rozwojowa . 12 (3): 403–413. doi : 10.1016/j.devcel.2007.01.012 . ISSN 1534-5807 . PMID 17336906 .
- ^ D'Lima NG, Ma J, Winkler L, Chu Q, Loh KH, Corpuz EO, Budnik BA, Lykke-Andersen J, Saghatelian A, Slavoff SA (luty 2017). „Ludzkie mikrobiałko, które oddziałuje z kompleksem otwierającym mRNA” . Biologia chemiczna natury . 13 (2): 174–180. doi : 10.1038/nchembio.2249 . PMC 5247292 . PMID 27918561 .
- ^ Ho, Lena; Tan, Shawn YX; Wee, Sheeno; Wu, Yixuan; Tan, Sam JC; Ramakryszna, Navin B.; Chng, Spokojny C.; Nama, Srikanth; Szczerbińska, Iwona; Szczerbińska, Iwona; Chan, Yun-Shen (01.10.2015). „ELABELA jest endogennym czynnikiem wzrostu, który podtrzymuje samoodnowę hESC poprzez ścieżkę PI3K/AKT” . Komórka Komórka Macierzysta . 17 (4): 435–447. doi : 10.1016/j.stem.2015.08.010 . ISSN 1875-9777 . PMID 26387754 .
- ^ Slavoff SA, Heo J, Budnik BA, Hanakahi LA, Saghatelian A (kwiecień 2014). „Ludzki polipeptyd kodowany w krótkiej otwartej ramce odczytu (sORF), który stymuluje łączenie końców DNA” . Czasopismo Chemii Biologicznej . 289 (16): 10950-7. doi : 10.1074/jbc.c113.533968 . PMC 4036235 . PMID 24610814 .
- ^ Guo B, Zhai D, Cabezas E, walijski K, Nouraini S, Satterthwait AC, Reed JC (maj 2003). „Peptyd ludzki hamuje apoptozę poprzez zakłócanie aktywacji Bax”. Natura . 423 (6938): 456–61. Kod bib : 2003Natur.423..456G . doi : 10.1038/nature01627 . PMID 12732850 . S2CID 4423176 .
- ^ Matsumoto A Pasut A Matsumoto M Yamashita R Fung J Monteleone E Saghatelian A Nakayama KI Clohessy JG Pandolfi PP (styczeń 2017). „mTORC1 i regeneracja mięśni są regulowane przez polipeptyd SPAR kodowany przez LINC00961”. Natura . 541 (7636): 228-232. Kod Bibcode : 2017Natur.541..228M . doi : 10.1038/nature21034 . PMID 28024296 . S2CID 205253245 .