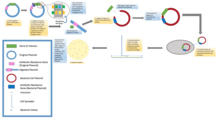
Klonowanie molekularne - Molecular cloning
Klonowanie molekularne to zestaw metod eksperymentalnych w biologii molekularnej, które są wykorzystywane do składania rekombinowanych cząsteczek DNA i kierowania ich replikacją w organizmach gospodarza . Użycie słowa klonowanie odnosi się do faktu, że metoda polega na replikacji jednej cząsteczki w celu wytworzenia populacji komórek o identycznych cząsteczkach DNA. Klonowanie molekularne na ogół wykorzystuje sekwencje DNA z dwóch różnych organizmów: gatunku, który jest źródłem klonowanego DNA oraz gatunku, który będzie służył jako żywy gospodarz do replikacji zrekombinowanego DNA. Metody klonowania molekularnego mają kluczowe znaczenie dla wielu współczesnych dziedzin współczesnej biologii i medycyny.
W konwencjonalnym eksperymencie klonowania molekularnego DNA do sklonowania uzyskuje się z organizmu będącego przedmiotem zainteresowania, a następnie traktuje się enzymami w probówce w celu wygenerowania mniejszych fragmentów DNA. Następnie te fragmenty łączy się następnie z wektorem DNA w celu wytworzenia rekombinowanych cząsteczek DNA. Zrekombinowany DNA jest następnie wprowadzany do organizmu gospodarza (zazwyczaj łatwego w hodowli, łagodnego szczepu laboratoryjnego bakterii E. coli ). Wygeneruje to populację organizmów, w której cząsteczki rekombinowanego DNA są replikowane wraz z DNA gospodarza. Ponieważ zawierają obce fragmenty DNA, są to mikroorganizmy transgeniczne lub genetycznie zmodyfikowane ( GMO ). Proces ten wykorzystuje fakt, że pojedyncza komórka bakteryjna może być indukowana do wchłonięcia i replikacji pojedynczej rekombinowanej cząsteczki DNA. Ta pojedyncza komórka może być następnie ekspandowana, aby wygenerować dużą ilość bakterii, z których każda zawiera kopie oryginalnej rekombinowanej cząsteczki. Tak więc zarówno uzyskana populacja bakterii, jak i rekombinowana cząsteczka DNA, są powszechnie określane jako „klony”. Ściśle mówiąc, rekombinowany DNA odnosi się do cząsteczek DNA, podczas gdy klonowanie molekularne odnosi się do metod eksperymentalnych stosowanych do ich składania. Pojawił się pomysł, że do plazmidu można wstawić różne sekwencje DNA i że te obce sekwencje zostaną przeniesione do bakterii i strawione jako część plazmidu. Oznacza to, że plazmidy te mogą służyć jako wektory do klonowania przenoszące geny.
Praktycznie każdą sekwencję DNA można sklonować i amplifikować, ale istnieją pewne czynniki, które mogą ograniczać powodzenie tego procesu. Przykładami sekwencji DNA, które są trudne do sklonowania, są odwrócone powtórzenia, miejsca początku replikacji, centromery i telomery. Istnieje również mniejsza szansa powodzenia przy wstawianiu sekwencji DNA o dużych rozmiarach. Inserty większe niż 10 kpz mają bardzo ograniczony sukces, ale bakteriofagi, takie jak bakteriofag λ, można zmodyfikować tak, aby z powodzeniem wstawić sekwencję do 40 kpz.
Historia
Przed latami 70. zrozumienie genetyki i biologii molekularnej było poważnie utrudnione przez niemożność izolowania i badania poszczególnych genów ze złożonych organizmów. Zmieniło się to dramatycznie wraz z pojawieniem się metod klonowania molekularnego. Mikrobiolodzy, chcąc zrozumieć mechanizmy molekularne, poprzez które bakterie ograniczały wzrost bakteriofagów, wyizolowali endonukleazy restrykcyjne , enzymy, które mogły rozszczepiać cząsteczki DNA tylko wtedy, gdy napotkano określone sekwencje DNA. Wykazali, że enzymy restrykcyjne rozszczepiają cząsteczki DNA o długości chromosomu w określonych miejscach i że określone sekcje większej cząsteczki można oczyszczać przez frakcjonowanie pod względem wielkości. Stosując drugi enzym, ligazę DNA , fragmenty wytworzone przez enzymy restrykcyjne można było łączyć w nowe kombinacje, zwane rekombinowanym DNA . Poprzez rekombinację interesujących segmentów DNA z wektorem DNA, takim jak bakteriofagi lub plazmidy, które naturalnie replikują się wewnątrz bakterii, można w kulturach bakteryjnych wytwarzać duże ilości oczyszczonych cząsteczek rekombinowanego DNA. Pierwsze rekombinowane cząsteczki DNA zostały wygenerowane i zbadane w 1972 roku.
Przegląd
Klonowanie molekularne wykorzystuje fakt, że struktura chemiczna DNA jest zasadniczo taka sama we wszystkich żywych organizmach. W związku z tym, jeśli jakikolwiek segment DNA z dowolnego organizmu zostanie wstawiony do segmentu DNA zawierającego sekwencje molekularne wymagane do replikacji DNA , a powstały zrekombinowany DNA zostanie wprowadzony do organizmu, z którego uzyskano sekwencje replikacyjne, wówczas obcy DNA zostanie zreplikowany wraz z DNA komórki gospodarza w organizmie transgenicznym .
Klonowanie molekularne jest podobne do reakcji łańcuchowej polimerazy (PCR), ponieważ umożliwia replikację sekwencji DNA. Podstawowa różnica między tymi dwiema metodami polega na tym, że klonowanie molekularne obejmuje replikację DNA w żywym mikroorganizmie, podczas gdy PCR replikuje DNA w roztworze in vitro , wolnym od żywych komórek.
Kroki
W standardowych eksperymentach z klonowaniem molekularnym klonowanie dowolnego fragmentu DNA obejmuje zasadniczo siedem etapów: (1) Wybór organizmu gospodarza i wektora do klonowania, (2) Przygotowanie wektorowego DNA, (3) Przygotowanie DNA do klonowania, (4) Stworzenie rekombinowanego DNA, (5) Wprowadzenie rekombinowanego DNA do organizmu gospodarza, (6) Selekcja organizmów zawierających rekombinowany DNA, (7) Przeszukiwanie klonów z pożądanymi wstawkami DNA i właściwościami biologicznymi.
Chociaż szczegółowe planowanie klonowania można wykonać w dowolnym edytorze tekstu, wraz z narzędziami online do np. projektowania starterów PCR, istnieje dedykowane oprogramowanie do tego celu. Oprogramowanie do tego celu to m.in. ApE [1] (open source), DNAStrider [2] (open source), Serial Cloner [3] (darmowy) oraz Collagene [4] (open source).
Warto zauważyć, że rosnąca wydajność i wierność platform syntezy DNA pozwala na coraz bardziej skomplikowane projekty w inżynierii molekularnej. Projekty te mogą obejmować bardzo długie nici nowej sekwencji DNA i/lub testować całe biblioteki jednocześnie, w przeciwieństwie do pojedynczych sekwencji. Te zmiany wprowadzają złożoność, która wymaga od projektu odejścia od płaskiej reprezentacji opartej na nukleotydach w kierunku wyższego poziomu abstrakcji. Przykładami takich narzędzi są GenoCAD , Teselagen [5] (bezpłatny dla środowiska akademickiego) czy GeneticConstructor [6] (bezpłatny dla środowiska akademickiego).
Wybór organizmu gospodarza i wektora do klonowania

Chociaż w użyciu jest bardzo duża liczba organizmów gospodarzy i wektorów do klonowania molekularnego, zdecydowana większość eksperymentów z klonowaniem molekularnym rozpoczyna się od laboratoryjnego szczepu bakterii E. coli ( Escherichia coli ) i wektora do klonowania plazmidu . Wektory E. coli i plazmidowe są powszechnie stosowane, ponieważ są wyrafinowane technicznie, wszechstronne, szeroko dostępne i oferują szybki wzrost organizmów rekombinowanych przy minimalnym sprzęcie. Jeśli DNA do sklonowania jest wyjątkowo duże (setki tysięcy do milionów par zasad), często wybierany jest sztuczny chromosom bakteryjny lub wektor sztucznych chromosomów drożdży .
Wyspecjalizowane aplikacje mogą wymagać wyspecjalizowanych systemów host-vector. Na przykład, jeśli eksperymentatorzy chcą zebrać określone białko z rekombinowanego organizmu, wówczas wybierany jest wektor ekspresyjny, który zawiera odpowiednie sygnały do transkrypcji i translacji w pożądanym organizmie gospodarza. Alternatywnie, jeśli pożądana jest replikacja DNA w różnych gatunkach (na przykład przeniesienie DNA z bakterii do roślin), wówczas można wybrać wektor z wielu gospodarzy (nazywany również wektorem wahadłowym ). W praktyce jednak eksperymenty z wyspecjalizowanym klonowaniem molekularnym zwykle rozpoczynają się od klonowania do plazmidu bakteryjnego, po którym następuje subklonowanie do wyspecjalizowanego wektora.
Niezależnie od zastosowanej kombinacji gospodarza i wektora, wektor prawie zawsze zawiera cztery segmenty DNA, które są krytycznie ważne dla jego funkcji i użyteczności eksperymentalnej:
- Początek replikacji DNA jest niezbędny, aby wektor (i połączone z nim sekwencje rekombinacyjne) mógł replikować się w organizmie gospodarza
- jedno lub więcej unikalnych miejsc rozpoznawanych przez endonukleazy restrykcyjne, które służą jako miejsca, w których można wprowadzić obce DNA
- wybierane genetyczny marker genu, które mogą być wykorzystane, aby umożliwić przeżycie komórek, które pobrały sekwencji wektora
- znacznik genów, które można stosować do przeszukiwania pod kątem komórek zawierających DNA obcego

Przygotowanie wektorowego DNA
Wektor do klonowania traktuje się endonukleazą restrykcyjną w celu odcięcia DNA w miejscu, w którym zostanie wstawiony obcy DNA. Enzym restrykcyjny wybiera się tak, aby wytworzył konfigurację w miejscu cięcia, która jest zgodna z końcami obcego DNA (patrz koniec DNA ). Zazwyczaj odbywa się to przez cięcie DNA wektora i obcego DNA tym samym enzymem restrykcyjnym, na przykład EcoRI . Większość nowoczesnych wektorów zawiera różne dogodne miejsca cięcia, które są unikalne w obrębie cząsteczki wektora (tak, że wektor może być cięty tylko w jednym miejscu) i są zlokalizowane w genie (często beta-galaktozydaza ), którego inaktywację można wykorzystać do rozróżnienia rekombinowany z organizmów nierekombinowanych na późniejszym etapie procesu. Aby poprawić stosunek organizmów zrekombinowanych do nierekombinowanych, rozszczepiony wektor można potraktować enzymem ( fosfataza alkaliczna ), który defosforyluje końce wektora. Cząsteczki wektora z defosforylowanymi końcami nie są zdolne do replikacji, a replikację można przywrócić tylko wtedy, gdy obcy DNA zostanie zintegrowany z miejscem cięcia.
Przygotowanie DNA do sklonowania
W celu klonowania genomowego DNA, klonowany DNA jest ekstrahowany z organizmu będącego przedmiotem zainteresowania. Można użyć praktycznie każdego źródła tkanki (nawet tkanek wymarłych zwierząt ), o ile DNA nie jest w znacznym stopniu zdegradowane. DNA jest następnie oczyszczany za pomocą prostych metod w celu usunięcia zanieczyszczających białek (ekstrakcja fenolem), RNA (rybonukleaza) i mniejszych cząsteczek (wytrącanie i/lub chromatografia). Metody reakcji łańcuchowej polimerazy (PCR) są często stosowane do amplifikacji określonych sekwencji DNA lub RNA ( RT-PCR ) przed klonowaniem molekularnym.
DNA do eksperymentów klonowania można również uzyskać z RNA przy użyciu odwrotnej transkryptazy ( klonowanie komplementarnego DNA lub cDNA) lub w postaci syntetycznego DNA ( synteza sztucznych genów ). Klonowanie cDNA jest zwykle stosowane do uzyskania klonów reprezentatywnych dla populacji mRNA interesujących komórek, podczas gdy syntetyczny DNA jest używany do uzyskania dowolnej dokładnej sekwencji określonej przez projektanta. Tak zaprojektowana sekwencja może być wymagana przy przenoszeniu genów przez kody genetyczne (na przykład z mitochrondrii do jądra) lub po prostu w celu zwiększenia ekspresji poprzez optymalizację kodonów .
Oczyszczony DNA jest następnie traktowany enzymem restrykcyjnym w celu wytworzenia fragmentów, których końce mogą być połączone z końcami wektora. Jeśli to konieczne, można dodać krótkie dwuniciowe segmenty DNA ( łączniki ) zawierające pożądane miejsca restrykcyjne, aby utworzyć końcowe struktury, które są kompatybilne z wektorem.
Tworzenie rekombinowanego DNA za pomocą ligazy DNA
Tworzenie zrekombinowanego DNA jest pod wieloma względami najprostszym etapem procesu klonowania molekularnego. DNA przygotowany z wektora i obcego źródła po prostu miesza się razem w odpowiednich stężeniach i wystawia na działanie enzymu ( ligazy DNA ), który kowalencyjnie łączy ze sobą końce. Ta reakcja łączenia jest często nazywana ligacją . Powstała mieszanina DNA zawierająca losowo połączone końce jest następnie gotowa do wprowadzenia do organizmu gospodarza.
Ligaza DNA rozpoznaje i działa tylko na końcach liniowych cząsteczek DNA, zwykle dając złożoną mieszaninę cząsteczek DNA o losowo połączonych końcach. Pożądane produkty (wektorowe DNA kowalencyjnie połączone z obcym DNA) będą obecne, ale inne sekwencje (np. obcy DNA połączony ze sobą, wektorowy DNA połączony ze sobą i kombinacje wyższego rzędu wektora i obcego DNA) są również zwykle obecne. Ta złożona mieszanina jest sortowana w kolejnych etapach procesu klonowania, po wprowadzeniu mieszaniny DNA do komórek.
Wprowadzenie rekombinowanego DNA do organizmu gospodarza
Mieszanina DNA, wcześniej manipulowana in vitro, jest przenoszona z powrotem do żywej komórki, zwanej organizmem gospodarza. Metody stosowane do wprowadzania DNA do komórek są różne, a nazwa tego etapu w procesie klonowania molekularnego będzie często zależeć od wybranej metody eksperymentalnej (np. transformacja , transdukcja , transfekcja , elektroporacja ).
Kiedy mikroorganizmy są w stanie pobierać i replikować DNA ze swojego lokalnego środowiska, proces ten nazywa się transformacją , a komórki, które są w stanie fizjologicznym, w którym mogą pobierać DNA, są uważane za kompetentne . W hodowli komórek ssaków, analogiczny proces wprowadzania DNA do komórek jest powszechnie nazywany transfekcją . Zarówno transformacja, jak i transfekcja zwykle wymagają przygotowania komórek poprzez specjalny reżim wzrostu i proces obróbki chemicznej, który będzie się różnił w zależności od konkretnych stosowanych gatunków i typów komórek.
Elektroporacja wykorzystuje impulsy elektryczne wysokiego napięcia do translokacji DNA przez błonę komórkową (i ścianę komórkową, jeśli jest obecna). W przeciwieństwie do tego, transdukcja obejmuje pakowanie DNA w cząstki pochodzące od wirusa i wykorzystanie tych wirusopodobnych cząstek do wprowadzenia zamkniętego w kapsułkach DNA do komórki w procesie przypominającym infekcję wirusową. Chociaż elektroporacja i transdukcja są metodami wysoce wyspecjalizowanymi, mogą być najskuteczniejszymi metodami przenoszenia DNA do komórek.
Selekcja organizmów zawierających sekwencje wektorowe
Niezależnie od zastosowanej metody, wprowadzenie rekombinowanego DNA do wybranego organizmu gospodarza jest zwykle procesem o niskiej wydajności; to znaczy, że tylko niewielka część komórek faktycznie przyjmie DNA. Eksperymentalni naukowcy radzą sobie z tym problemem poprzez etap sztucznej selekcji genetycznej, w którym komórki, które nie wchłonęły DNA, są selektywnie zabijane, i tylko te komórki, które potrafią aktywnie replikować DNA zawierające gen markera selekcyjnego kodowany przez wektor, są w stanie przetrwać.
Gdy jako organizmy gospodarza stosuje się komórki bakteryjne, markerem selekcyjnym jest zwykle gen, który nadaje oporność na antybiotyk , który w innym przypadku zabiłby komórki, zazwyczaj ampicylinę . Komórki niosące plazmid przeżyją po ekspozycji na antybiotyk, podczas gdy te, które nie przyjęły sekwencji plazmidu, umrą. Gdy stosuje się komórki ssaków (np. komórki ludzkie lub mysie), stosuje się podobną strategię, z wyjątkiem tego, że gen markerowy (w tym przypadku typowo kodowany jako część kasety kanMX ) nadaje oporność na antybiotyk Genetycynę .
Badanie przesiewowe pod kątem klonów o pożądanych wstawkach DNA i właściwościach biologicznych
Nowoczesne bakteryjne wektory do klonowania (np. pUC19 i późniejsze pochodne, w tym wektory pGEM) wykorzystują niebiesko-biały system przesiewowy do odróżnienia kolonii (klonów) komórek transgenicznych od tych, które zawierają wektor macierzysty (tj. wektor DNA bez wstawionej sekwencji rekombinowanej). W tych wektorach obcy DNA jest wstawiany do sekwencji, która koduje zasadniczą część beta-galaktozydazy , enzymu, którego aktywność powoduje tworzenie kolonii o niebieskim zabarwieniu na pożywce hodowlanej, która jest używana do tej pracy. Wstawienie obcego DNA do sekwencji kodującej beta-galaktozydazę wyłącza funkcję enzymu, tak że kolonie zawierające transformowany DNA pozostają bezbarwne (białe). Dlatego eksperymentatorzy są w stanie łatwo zidentyfikować i przeprowadzić dalsze badania nad transgenicznymi klonami bakteryjnymi, ignorując te, które nie zawierają rekombinowanego DNA.
Całkowita populacja pojedynczych klonów uzyskanych w eksperymencie klonowania molekularnego jest często nazywana biblioteką DNA . Biblioteki mogą być bardzo złożone (jak klonowanie całego genomowego DNA z organizmu) lub stosunkowo proste (jak przenoszenie wcześniej sklonowanego fragmentu DNA do innego plazmidu), ale prawie zawsze konieczne jest zbadanie kilku różnych klonów, aby mieć pewność otrzymanie pożądanego konstruktu DNA. Można to osiągnąć za pomocą bardzo szerokiego zakresu metod eksperymentalnych, w tym zastosowania hybrydyzacji kwasów nukleinowych , sond przeciwciał , reakcji łańcuchowej polimerazy , analizy fragmentów restrykcyjnych i/lub sekwencjonowania DNA .
Aplikacje
Klonowanie molekularne zapewnia naukowcom zasadniczo nieograniczoną liczbę pojedynczych segmentów DNA pochodzących z dowolnego genomu. Materiał ten może być wykorzystywany do wielu celów, w tym zarówno w podstawowych, jak i stosowanych naukach biologicznych. Poniżej podsumowano kilka ważniejszych zastosowań.
Organizacja genomu i ekspresja genów
Klonowanie molekularne doprowadziło bezpośrednio do wyjaśnienia pełnej sekwencji DNA genomów bardzo dużej liczby gatunków oraz do zbadania różnorodności genetycznej w obrębie poszczególnych gatunków, co zostało wykonane głównie poprzez określenie sekwencji DNA dużej liczby losowo sklonowane fragmenty genomu i złożenie nakładających się sekwencji.
Na poziomie poszczególnych genów klony molekularne stosuje się do wytwarzania sond , które są wykorzystywane do badania, jak geny są wyrażone i sposób, że ekspresja jest związana z innych procesów biologicznych, w tym środowiska metabolicznego sygnałów zewnątrzkomórkowej, rozwoju, uczenia się, starzenia i śmierć komórki. Sklonowane geny mogą również dostarczyć narzędzi do badania funkcji biologicznej i znaczenia poszczególnych genów, umożliwiając badaczom inaktywację genów lub dokonywanie bardziej subtelnych mutacji przy użyciu mutagenezy regionalnej lub mutagenezy ukierunkowanej . Geny sklonowane do wektorów ekspresyjnych do klonowania funkcjonalnego zapewniają środki do przeszukiwania genów na podstawie funkcji wyrażanego białka.
Produkcja białek rekombinowanych
Uzyskanie klonu molekularnego genu może prowadzić do rozwoju organizmów wytwarzających produkt białkowy sklonowanych genów, określany jako białko rekombinowane. W praktyce często trudniej jest opracować organizm, który wytwarza aktywną formę rekombinowanego białka w pożądanych ilościach niż klonowanie genu. Dzieje się tak, ponieważ sygnały molekularne do ekspresji genów są złożone i zmienne, a fałdowanie, stabilność i transport białek mogą być bardzo trudne.
Wiele użytecznych białek jest obecnie dostępnych jako produkty rekombinowane . Należą do nich: (1) medycznie użyteczne białka, których podanie może skorygować wadliwy lub słabo wyrażony gen (np. rekombinowany czynnik VIII , niedobór czynnika krzepnięcia krwi w niektórych postaciach hemofilii oraz rekombinowana insulina , stosowana w leczeniu niektórych postaci cukrzycy ) , (2) białka, które można podawać w sytuacjach zagrożenia życia (np. tkankowy aktywator plazminogenu , stosowany w leczeniu udarów), (3) szczepionki podjednostkowe rekombinowane, w których oczyszczone białko można wykorzystać do uodporniania pacjentów przeciwko chorobom zakaźnym , bez narażania ich na sam czynnik zakaźny (np. szczepionka przeciwko wirusowemu zapaleniu wątroby typu B ) oraz (4) rekombinowane białka jako standardowy materiał do diagnostycznych testów laboratoryjnych.
Organizmy transgeniczne
Po scharakteryzowaniu i zmanipulowaniu w celu dostarczenia sygnałów do odpowiedniej ekspresji, sklonowane geny można wprowadzić do organizmów, tworząc organizmy transgeniczne, zwane również organizmami zmodyfikowanymi genetycznie (GMO). Chociaż większość GMO jest generowana na potrzeby podstawowych badań biologicznych (patrz na przykład myszy transgeniczne ), wiele GMO zostało opracowanych do użytku komercyjnego, począwszy od zwierząt i roślin produkujących leki lub inne związki ( farmowanie ), upraw odpornych na herbicydy rośliny i fluorescencyjne ryby tropikalne ( GloFish ) do domowej rozrywki.
Terapia genowa
Terapia genowa polega na dostarczaniu funkcjonalnego genu do komórek pozbawionych tej funkcji w celu skorygowania zaburzenia genetycznego lub choroby nabytej. Terapię genową można ogólnie podzielić na dwie kategorie. Pierwszym z nich jest przekształcenie komórek zarodkowych, czyli plemników lub komórek jajowych, co skutkuje trwałą zmianą genetyczną całego organizmu i kolejnych pokoleń. Ta „terapia genowa linii zarodkowej” jest uważana przez wielu za nieetyczną u ludzi. Drugi rodzaj terapii genowej, „terapia genowa komórek somatycznych”, jest analogiczny do przeszczepu narządu. W tym przypadku celem bezpośredniego leczenia lub usunięcia tkanki, dodania genu lub genów terapeutycznych w laboratorium i powrotu leczonych komórek pacjentowi, jest jedna lub więcej określonych tkanek. Próby kliniczne terapii genowej komórek somatycznych rozpoczęły się pod koniec lat 90., głównie w leczeniu nowotworów oraz chorób krwi, wątroby i płuc.
Pomimo ogromnego rozgłosu i obietnic, historia ludzkiej terapii genowej charakteryzuje się stosunkowo ograniczonym sukcesem. Efekt wprowadzenia genu do komórek często sprzyja jedynie częściowemu i/lub przejściowemu złagodzeniu objawów leczonej choroby. Niektórzy pacjenci biorący udział w badaniu terapii genowej ponieśli negatywne konsekwencje samego leczenia, w tym zgony. W niektórych przypadkach działania niepożądane wynikają z uszkodzenia podstawowych genów w genomie pacjenta przez inaktywację insercyjną. W innych wektory wirusowe stosowane w terapii genowej zostały skażone wirusem zakaźnym. Niemniej jednak, terapia genowa jest nadal uważana za obiecującą przyszłą dziedzinę medycyny i jest to dziedzina, w której istnieje znaczny poziom działalności badawczo-rozwojowej.
Bibliografia
Dalsza lektura
- Matsumura, Ichiro (wrzesień 2015). „Dlaczego Johnny nie może klonować: typowe pułapki i niezbyt powszechne rozwiązania” . Biotechniki . 53 (3): IV–XIII. doi : 10.2144/000114324 . PMID 26345511 . Zarchiwizowane od oryginału dnia 2015-09-16 . Pobrano 2 lutego 2016 .
Zewnętrzne linki
|
Zasoby biblioteczne dotyczące klonowania molekularnego |
-
 Multimedia związane z klonowaniem molekularnym w Wikimedia Commons
Multimedia związane z klonowaniem molekularnym w Wikimedia Commons