Peptydomimetyk - Peptidomimetic
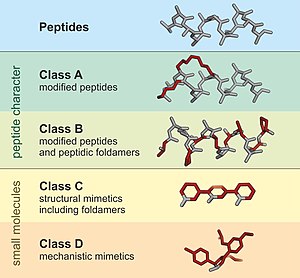
Peptydomimetyczny jest mała białkowej łańcucha przeznaczony do naśladowania peptydu . Zwykle powstają one albo w wyniku modyfikacji istniejącego peptydu, albo przez zaprojektowanie podobnych systemów, które naśladują peptydy, takie jak peptoidy i β-peptydy . Niezależnie od podejścia, zmieniona struktura chemiczna jest zaprojektowana tak, aby korzystnie dostosować właściwości molekularne, takie jak stabilność lub aktywność biologiczna . Może to odgrywać rolę w rozwoju związków podobnych do leków z istniejących peptydów. Modyfikacje te obejmują zmiany w peptydzie, które nie wystąpią naturalnie (takie jak zmienione szkielety i włączenie nienaturalnych aminokwasów). Na podstawie ich podobieństwa do peptydu prekursorowego, peptydomimetyki można podzielić na cztery klasy (A – D), gdzie A wykazuje największe podobieństwo, a D najmniej. Klasy A i B obejmują rusztowania podobne do peptydów, podczas gdy klasy C i D obejmują małe cząsteczki (ryc. 1).
Peptydy
Składacze
D-peptydy
Peptyd D to niewielka sekwencja D-aminokwasów . Ponieważ rybosomy są specyficzne dla L-aminokwasów, D-peptydy rzadko występują naturalnie w organizmach i nie są łatwo trawione ani degradowane. Peptydomimetyki D-peptydowe to D-peptydy zaprojektowane tak, aby naśladować naturalne peptydy L, które powszechnie mają właściwości terapeutyczne.
Właściwości D-peptydów

Po umieszczeniu w niechiralnym rozpuszczalniku, takim jak woda, peptydy D, a także większe białka D polipeptydu, mają podobne, ale lustrzane właściwości do peptydów L i białek L o identycznych sekwencjach. Jeśli białko L nie wymaga chaperonu lub kofaktora strukturalnego do fałdowania, jego białko enancjomeru D powinno mieć konformację lustrzanego odbicia w stosunku do białka L (Figura 2). Enzym D powinien działać na substraty o odwrotnej chiralności w porównaniu z enzymem L o tej samej sekwencji. Podobnie, jeśli peptyd L wiąże się z białkiem L, ich odpowiedniki w postaci peptydu D i białka D powinny wiązać się ze sobą w sposób lustrzany.
Peptydy D mają również właściwości, które czynią je atrakcyjnymi jako leki. Peptydy D są mniej podatne na rozkład w żołądku lub wewnątrz komórek na drodze proteolizy . Leki D-peptydowe można zatem przyjmować doustnie i są skuteczne przez dłuższy czas. Peptydy D są łatwe do syntezy w porównaniu z wieloma innymi lekami. W niektórych przypadkach peptydy D mogą wykazywać niską odpowiedź immunogenną .
Metody projektowania D-peptydów
Projekt ret
Peptyd L ma trzy sekwencje analogowe (Figura 3) zbudowane z aminokwasów L i D: enancjomer D lub inwersopeptyd o tej samej sekwencji, ale złożony z D-aminokwasów i konformacji lustrzanej; retropeptyd, składający się z tej samej sekwencji aminokwasów L, ale w odwrotnej kolejności; oraz peptyd retro-inverso lub D-retro-enancjomer, składający się z D-aminokwasów w odwróconej sekwencji.
Podczas gdy L-peptyd i jego D-enancjomer są swoimi lustrzanymi strukturami, L-retro-peptyd jest lustrzanym odbiciem D-retro-inwerso-peptydu. Z drugiej strony, peptyd L i D-retro-inwerso-peptyd mają podobny układ łańcuchów bocznych, chociaż ich grupy karboksylowe i aminowe wskazują przeciwne kierunki. W przypadku małych peptydów, które nie zależą od drugorzędowej struktury do wiązania, peptyd L i jego D-retro-inverso-peptyd prawdopodobnie mają podobne powinowactwo wiązania z docelowym białkiem L.

Wyświetlanie fagowe w lustrzanym odbiciu
Prezentacja fagowa to technika przeszukiwania dużych bibliotek peptydów pod kątem wiązania się z docelowym białkiem. W prezentacji fagowej sekwencja DNA kodująca potencjalny lek-peptyd jest łączona z genem płaszcza białkowego bakteriofagów i wprowadzana do wektora. Różnorodność można wprowadzić do peptydu przez mutagenezę . Peptydy otoczki białkowej są następnie eksprymowane i oczyszczane oraz nakładane na powierzchnię unieruchomionych celów białkowych. Powierzchnia jest następnie wypłukiwana w celu usunięcia niewiążących peptydów, podczas gdy pozostałe peptydy wiążące są eluowane.
Prezentacja na fagu lustrzanym jest podobną metodą, którą można zastosować do przeszukiwania dużych bibliotek peptydów D, które wiążą się z docelowymi białkami L. Dokładniej, ponieważ D-peptydy nie mogą ulegać ekspresji w bakteriofagach, lustrzane odbicia fagowe przesiewają peptydy L, które wiążą się z unieruchomionymi białkami D, które zostały wcześniej zsyntetyzowane chemicznie . Ze względu na lustrzane właściwości peptydów D, enancjomer D peptydu L, który wiąże się z białkiem D, wiąże się z białkiem L.
Wyświetlanie fagowe w lustrzanym odbiciu ma jednak dwie wady w porównaniu z prezentacją fagową. Docelowe białka D muszą być syntetyzowane chemicznie, co jest zwykle procesem kosztownym i czasochłonnym. Ponadto białko docelowe nie może wymagać kofaktora lub białka opiekuńczego do fałdowania, w przeciwnym razie chemicznie zsyntetyzowane białko D nie będzie fałdować się do docelowej, lustrzanej struktury.

Podobieństwo strukturalne
Peptydu o strukturze drugorzędowej nie można naśladować przez jego retro-odwrotność, ponieważ łączenie w odwrotnej kolejności łamie wiele interakcji szkieletowych istotnych dla struktury drugorzędowej. Podejściem do naśladowania tych peptydów jest poszukiwanie podobnych (łańcuchów bocznych) struktur w kopii lustrzanej Protein Data Bank dla elementów strukturyzowanych, a następnie łączenie sekcji przez retro-odwrócone wersje pętli znajdujących się w oryginalnym białku.
Małe cząsteczki
Przykłady
Podejścia peptydomimetyczne zostały wykorzystane do zaprojektowania małych cząsteczek, które selektywnie zabijają komórki rakowe, podejście znane jako chemioterapia celowana , poprzez indukowanie zaprogramowanej śmierci komórki w procesie zwanym apoptozą . Poniższe dwa przykłady naśladują białka zaangażowane w kluczowe interakcje białko-białko, które reaktywują szlak apoptotyczny w raku, ale robią to za pomocą odrębnych mechanizmów.
W 2004 roku Walensky i współpracownicy donieśli o stabilizowanym alfa-helikalnym peptydzie, który naśladuje proapoptotyczne białka wyłącznie BH3, takie jak BID i BAD . Ta cząsteczka została zaprojektowana w celu stabilizacji natywnej struktury helikalnej poprzez utworzenie makrocyklu między łańcuchami bocznymi, które nie biorą udziału w wiązaniu. Proces ten, zwany staplowaniem peptydów , wykorzystuje nienaturalne aminokwasy w celu ułatwienia makrocyklizacji poprzez metatezę olefin z zamknięciem pierścienia . W tym przypadku zidentyfikowano zszytą helisę BH3, która specyficznie aktywuje mitochondrialny szlak apoptozy poprzez antagonizowanie sekwestracji białek wyłącznie BH3 przez białka antyapoptotyczne (np. Bcl-2 , patrz także wewnętrzne i zewnętrzne induktory apoptozy). Ta cząsteczka hamowała wzrost ludzkiej białaczki w mysim modelu heteroprzeszczepu .
Również w 2004 roku Harran i współpracownicy donieśli o dimerycznej małej cząsteczce, która naśladuje proapoptotyczne białko Smac (patrz regulacja mitochondrialna w apoptozie). Ta cząsteczka naśladuje N-końcowy motyw liniowy Ala-Val-Pro-Ile. Wyjątkowo struktura dimeryczna tego peptydomimetyku doprowadziła do znacznego wzrostu aktywności w porównaniu z analogicznym monomerem. Ta kooperacyjność wiązania wynika ze zdolności cząsteczki do naśladowania struktury homodimerycznej Smac, co jest funkcjonalnie ważne dla reaktywacji kaspaz. Mimetyki Smac tego typu mogą uwrażliwiać szereg niedrobnokomórkowych komórek raka płuc na konwencjonalne chemioterapeutyki (np. gemcytabinę , winorelbinę ) zarówno in vitro, jak iw mysich modelach przeszczepów ksenogenicznych.
Heterocykle są często używane do naśladowania wiązania amidowego peptydów. Na przykład tiazole znajdują się w naturalnie występujących peptydach i są wykorzystywane przez naukowców do naśladowania wiązania amidowego peptydu.
Zobacz też
- Apoptoza
- Beta-peptyd
- Nowotwór
- Kliknięty polimer peptydowy
- Depsipeptyd
- Rozszerzony kod genetyczny
- Składacze
- Aminokwasy nieproteogenne