Badania przesiewowe dwuhybrydowe - Two-hybrid screening
Dwuhybrydowe badanie przesiewowe (pierwotnie znane jako drożdżowy system dwuhybrydowy lub Y2H ) to technika biologii molekularnej stosowana do wykrywania interakcji białko-białko (PPI) i interakcji białko-DNA poprzez badanie interakcji fizycznych (takich jak wiązanie) między dwoma białkami lub odpowiednio pojedyncze białko i cząsteczka DNA .
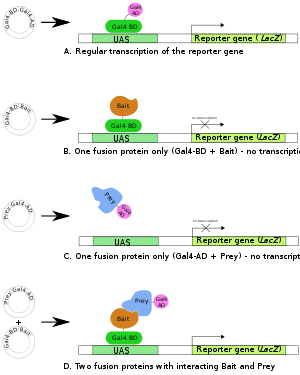
Przesłanką testu jest aktywacja genu ( genów) reporterowych w dół rzeki przez wiązanie czynnika transkrypcyjnego z sekwencją aktywującą w górę (UAS). W przypadku badań przesiewowych dwuhybrydowych czynnik transkrypcyjny jest podzielony na dwa oddzielne fragmenty, zwane domeną wiążącą DNA (DBD lub często również w skrócie BD) i domeną aktywującą (AD). BD jest domeną odpowiedzialną za wiązanie z UAS, a AD jest domeną odpowiedzialną za aktywację transkrypcji . Y2H jest zatem testem komplementacji fragmentów białka .
Historia
Technika ta, zapoczątkowana przez Stanleya Fieldsa i Ok-Kyu Songa w 1989 roku, została pierwotnie zaprojektowana do wykrywania interakcji białko-białko przy użyciu aktywatora transkrypcji Gal4 drożdży Saccharomyces cerevisiae . Gal4 aktywowane białko transkrypcję genu zaangażowanego w wykorzystania galaktozy, która stanowiła podstawę selekcji. Od tego czasu ta sama zasada została dostosowana do opisania wielu alternatywnych metod, w tym takich, które wykrywają interakcje białko-DNA lub interakcje DNA-DNA , a także metod wykorzystujących różne organizmy gospodarza, takie jak Escherichia coli lub komórki ssaków zamiast drożdży.
Podstawowa przesłanka
Kluczem do przesiewania dwuhybrydowego jest to, że w większości eukariotycznych czynników transkrypcyjnych domeny aktywujące i wiążące są modularne i mogą funkcjonować blisko siebie bez bezpośredniego wiązania. Oznacza to, że chociaż czynnik transkrypcyjny jest podzielony na dwa fragmenty, nadal może aktywować transkrypcję, gdy te dwa fragmenty są połączone pośrednio.
Najpopularniejszym podejściem do badań przesiewowych jest test dwuhybrydowy na drożdżach. W tym podejściu badacz wie, gdzie znajduje się każda zdobycz na użytej pożywce (płytkach agarowych). W ciągu ostatniej dekady zbadano miliony potencjalnych interakcji w kilku organizmach przy użyciu wysokowydajnych systemów przesiewowych (często z wykorzystaniem robotów), a ponad tysiące interakcji wykryto i sklasyfikowano w bazach danych jako BioGRID . System ten często wykorzystuje genetycznie zmodyfikowany szczep drożdży, w którym brakuje biosyntezy niektórych składników odżywczych (zwykle aminokwasów lub kwasów nukleinowych ). Gdy są hodowane na podłożach pozbawionych tych składników odżywczych, drożdże nie przetrwają. Ten zmutowany szczep drożdży można wytworzyć tak, aby zawierał obcy DNA w postaci plazmidów . W dwuhybrydowym badaniu przesiewowym drożdży, oddzielne plazmidy przynęty i zdobyczy są jednocześnie wprowadzane do zmutowanego szczepu drożdży lub stosuje się strategię kojarzenia, aby uzyskać oba plazmidy w jednej komórce gospodarza.
Drugim podejściem wysokoprzepustowym jest podejście do przeszukiwania bibliotek. W tej konfiguracji komórki kryjące przynętę i zdobycz kojarzą się w losowej kolejności. Po kojarzeniu i selekcji komórek, które przeżyły na pożywce selekcyjnej, naukowiec przeprowadzi sekwencjonowanie wyizolowanych plazmidów, aby zobaczyć, która zdobycz (sekwencja DNA) wchodzi w interakcję z użytą przynętą. Podejście to ma niższy współczynnik odtwarzalności i daje większą liczbę wyników fałszywie dodatnich w porównaniu z podejściem matrycowym.
Plazmidy są konstruowane w celu wytworzenia produktu białkowego, w którym fragment domeny wiążącej DNA (BD) jest połączony z białkiem, podczas gdy inny plazmid jest konstruowany w celu wytworzenia produktu białkowego, w którym fragment domeny aktywacji (AD) jest połączony z innym białkiem. Białko połączone z BD może być określane jako białko przynęty i jest zazwyczaj znanym białkiem, którego badacz używa do identyfikacji nowych partnerów wiążących. Białko połączone z AD może być określane jako białko zdobyczne i może być albo pojedynczym znanym białkiem albo biblioteką znanych lub nieznanych białek. W tym kontekście biblioteka może składać się ze zbioru sekwencji kodujących białka, które reprezentują wszystkie białka ulegające ekspresji w określonym organizmie lub tkance, lub może być wytworzona przez syntezę losowych sekwencji DNA. Niezależnie od źródła, są one następnie włączane do sekwencji kodującej białko plazmidu, który jest następnie transfekowany do komórek wybranych do metody przesiewowej. Ta technika, przy użyciu biblioteki, zakłada, że każda komórka jest transfekowana nie więcej niż jednym plazmidem, a zatem każda komórka ostatecznie wyraża nie więcej niż jeden składnik z biblioteki białkowej.
Jeśli białka przynęty i ofiary oddziałują (tj. wiążą), wtedy AD i BD czynnika transkrypcyjnego są połączone pośrednio, zbliżając AD do miejsca rozpoczęcia transkrypcji i może wystąpić transkrypcja genu(ów) reporterowego(ych). Jeśli te dwa białka nie oddziałują, nie ma transkrypcji genu reporterowego. W ten sposób udana interakcja między fuzyjnym białkiem jest powiązana ze zmianą fenotypu komórki.
Wyzwanie polegające na oddzieleniu komórek, które wyrażają białka, które wchodzą w interakcję z ich odpowiednikami białkami fuzyjnymi od tych, które tego nie robią, omówiono w następnej sekcji.
Domeny stałe
W każdym badaniu, niektóre z domen białkowych, te badane, będą zmieniane zgodnie z celami badania, podczas gdy inne domeny, te, które same nie są badane, będą utrzymywane na stałym poziomie. Na przykład w badaniu dwuhybrydowym w celu wybrania domen wiążących DNA, domena wiążąca DNA, BD, będzie się zmieniać, podczas gdy dwa oddziałujące białka, przynęta i ofiara, muszą być utrzymywane na stałym poziomie, aby utrzymać silne wiązanie między BD i AD. Istnieje wiele domen, z których należy wybrać BD, przynętę i zdobycz oraz AD, jeśli mają pozostać niezmienione. W badaniach interakcji białko-białko BD można wybrać spośród wielu domen silnie wiążących DNA, takich jak Zif268 . Częstym wyborem domen przynęty i zdobyczy są odpowiednio reszty 263-352 drożdży Gal11P z mutacją N342V i reszty 58-97 drożdży Gal4. Domeny te można stosować zarówno w technikach selekcji opartych na drożdżach, jak i bakteriach i wiadomo, że silnie się ze sobą wiążą.
Wybrana AD musi być w stanie aktywować transkrypcję genu reporterowego przy użyciu własnej maszynerii transkrypcyjnej komórki. W związku z tym różnorodność AD dostępnych do stosowania w technikach opartych na drożdżach może nie być odpowiednia do zastosowania w ich analogach opartych na bakteriach. AD pochodzące z wirusa opryszczki pospolitej, VP16 i drożdżowe Gal4 AD zostały z powodzeniem zastosowane w drożdżach, podczas gdy część podjednostki a polimerazy RNA E. coli została wykorzystana w metodach opartych na E. coli .
Podczas gdy silnie aktywujące domeny mogą pozwolić na większą wrażliwość na słabsze interakcje, odwrotnie, słabszy AD może zapewnić większą rygorystyczność.
Konstrukcja plazmidów ekspresyjnych
Aby przeprowadzić analizę dwuhybrydową lub jedną z jej pochodnych technik, do komórki gospodarza należy wprowadzić pewną liczbę zmodyfikowanych sekwencji genetycznych. Rozważania i metody stosowane w konstrukcji i dostarczaniu tych sekwencji różnią się w zależności od potrzeb testu i organizmu wybranego jako tło eksperymentu.
Istnieją dwie szerokie kategorie bibliotek hybrydowych: biblioteki losowe i biblioteki oparte na cDNA. Biblioteka cDNA, składa się cDNA wytwarza się przez odwrotną transkrypcję mRNA pobierana od konkretnych komórkach typu komórki. Ta biblioteka może być zligowana do konstruktu tak, aby była przyłączona do BD lub AD używanej w teście. Losowa biblioteka wykorzystuje długości DNA o losowej sekwencji zamiast tych odcinków cDNA. Istnieje szereg metod wytwarzania tych losowych sekwencji, w tym mutageneza kasetowa . Niezależnie od źródła biblioteki DNA, jest ona ligowana w odpowiednim miejscu w odpowiednim plazmidzie/fagemidzie przy użyciu odpowiednich endonukleaz restrykcyjnych .
Uwagi dotyczące E. coli
Umieszczając białka hybrydowe pod kontrolą promotorów lac indukowanych przez IPTG , są one wyrażane tylko na pożywkach uzupełnionych IPTG. Ponadto, włączając różne geny oporności na antybiotyki w każdym konstrukcie genetycznym, można łatwo zapobiegać wzrostowi nieprzekształconych komórek poprzez hodowlę na podłożach zawierających odpowiednie antybiotyki. Jest to szczególnie ważne w przypadku metod selekcji licznikowej, w których do przeżycia komórki potrzebny jest brak interakcji.
Gen reporterowy można wstawić do genomu E. coli , najpierw wstawiając go do episomu , rodzaju plazmidu zdolnego do włączania się do genomu komórki bakteryjnej z liczbą kopii około jednej na komórkę.
Hybrydowe fagemidy ekspresyjne można poddać elektroporacji do komórek E. coli XL-1 Blue, które po amplifikacji i infekcji fagiem pomocniczym VCS-M13 dadzą zapas faga z biblioteki. Każdy z tych fagów będzie zawierał jednego jednoniciowego członka biblioteki fagemidowej.
Odzyskiwanie informacji o białkach
Po dokonaniu selekcji należy określić pierwotną strukturę białek, które wykazują odpowiednią charakterystykę. Osiąga się to poprzez odzyskanie sekwencji kodujących białko (tak jak oryginalnie wstawione) z komórek wykazujących odpowiedni fenotyp.
E coli
Fagemid stosowany do transformacji komórek E. coli może być „uratowany” z wybranych komórek przez zainfekowanie ich fagiem pomocniczym VCS-M13. Powstałe cząstki faga, które są wytwarzane, zawierają jednoniciowe fagemidy i są wykorzystywane do infekowania komórek XL-1 Blue. Dwuniciowe fagmidy są następnie zbierane z tych komórek XL-1 Blue, zasadniczo odwracając proces stosowany do wytworzenia oryginalnej biblioteki faga. Na koniec sekwencje DNA są określane przez sekwencjonowanie dideoksy .
Kontrolowanie czułości
W Escherichia coli pochodzące z A tet R represorowe mogą być stosowane zgodnie z konwencjonalnym genu reporterowego i mogą być sterowane przez tetracyklinę lub doxicycline (inhibitory tet R). Zatem ekspresja Tet-R jest kontrolowana przez standardowy system dwuhybrydowy, ale Tet-R z kolei kontroluje (represjonuje) ekspresję wcześniej wspomnianego reportera, takiego jak HIS3 , poprzez jego promotor Tet-R. Tetracyklinę lub jej pochodne można następnie zastosować do regulacji czułości układu wykorzystującego Tet-R.
Czułość można również kontrolować zmieniając zależność komórek od ich genów reporterowych. Na przykład, może na to wpływać zmiana stężenia histydyny w pożywce wzrostowej dla komórek zależnych od his3 i zmiana stężenia streptomycyny dla komórek zależnych od aadA . Zależność od genu selekcyjnego można również kontrolować przez zastosowanie inhibitora genu selekcyjnego w odpowiednim stężeniu. Na przykład 3-amino-1,2,4-triazol (3-AT) jest konkurencyjnym inhibitorem produktu genu HIS3 i może być stosowany do miareczkowania minimalnego poziomu ekspresji HIS3 wymaganego do wzrostu na pożywkach z niedoborem histydyny.
Czułość można również modulować przez zmianę liczby sekwencji operatorowych w DNA reporterowym.
Białka niefuzyjne
Trzecie, niefuzyjne białko może być koeksprymowane z dwoma białkami fuzyjnymi. W zależności od badania, trzecie białko może modyfikować jedno z białek fuzyjnych lub pośredniczyć lub zakłócać ich interakcję.
Koekspresja trzeciego białka może być konieczna do modyfikacji lub aktywacji jednego lub obu białek fuzyjnych. Na przykład S. cerevisiae nie posiada endogennej kinazy tyrozynowej. Jeśli badanie dotyczy białka, które wymaga fosforylacji tyrozyny, kinaza musi być dostarczana w postaci genu kinazy tyrozynowej.
Białko niefuzyjne może pośredniczyć w oddziaływaniu przez jednoczesne wiązanie obu białek fuzyjnych, jak w przypadku zależnej od liganda dimeryzacji receptora.
W przypadku białka z oddziałującym partnerem, jego funkcjonalną homologię z innymi białkami można ocenić dostarczając trzecie białko w postaci niefuzyjnej, które następnie może konkurować lub nie z białkiem fuzyjnym o jego partnera wiążącego. Wiązanie pomiędzy trzecim białkiem a drugim białkiem fuzyjnym przerwie tworzenie kompleksu aktywującego ekspresję reportera, a tym samym zmniejszy ekspresję reportera, prowadząc do odróżniającej zmiany w fenotypie.
Drożdże Split-ubikwitynowe dwuhybrydowe
Jednym z ograniczeń klasycznych dwuhybrydowych badań przesiewowych drożdży jest to, że są one ograniczone do białek rozpuszczalnych. Dlatego niemożliwe jest ich wykorzystanie do badania oddziaływań białko-białko między nierozpuszczalnymi integralnymi białkami błonowymi . System split-ubikwityny zapewnia metodę przezwyciężenia tego ograniczenia. W układzie rozszczepionym ubikwityny dwa integralne białka błonowe, które mają być badane, są połączone z dwoma różnymi ugrupowaniami ubikwityny : C-końcową cząstką ubikwityny („Cub”, reszty 35–76) i N-końcową cząstką ubikwityny („Nub”, pozostałości 1-34)). Te połączone białka nazywane są odpowiednio przynętą i zdobyczą. Oprócz połączenia z integralnym białkiem błonowym, ugrupowanie Cub jest również połączone z czynnikiem transkrypcyjnym (TF), który może być odcięty przez proteazy specyficzne dla ubikwityny . Po interakcji przynęta-ofiara, ugrupowania Nub i Cub łączą się, odtwarzając rozszczepioną ubikwitynę. Odtworzona cząsteczka split-ubikwityny jest rozpoznawana przez proteazy specyficzne dla ubikwityny, które odcinają czynnik transkrypcyjny, umożliwiając indukowanie transkrypcji genów reporterowych .
Fluorescencyjny test dwuhybrydowy
Zolghadr i współpracownicy przedstawili fluorescencyjny system dwuhybrydowy, który wykorzystuje dwa białka hybrydowe, które są połączone z różnymi białkami fluorescencyjnymi, jak również z LacI, represorem lac . Struktura białek fuzyjnych wygląda następująco: FP2-LacI-przynęta i FP1-zdobycz, gdzie białka przynęty i zdobyczy oddziałują i zbliżają białka fluorescencyjne (FP1 = GFP , FP2= mCherry ) w miejscu wiązania LacI białko w genomie komórki gospodarza. System może być również używany do badań przesiewowych pod kątem inhibitorów oddziaływań białko-białko.
Enzymatyczne systemy dwuhybrydowe: KISS
Podczas gdy oryginalny system Y2H wykorzystywał odtworzony czynnik transkrypcyjny, inne systemy wytwarzają aktywność enzymatyczną w celu wykrywania PPI. Na przykład KInase Substrate Sensor ("KISS"), jest podejściem dwuhybrydowym ssaków, zaprojektowanym do mapowania wewnątrzkomórkowych PPI. Tutaj białko przynęty jest połączone z częścią TYK2 zawierającą kinazę, a ofiara jest sprzęgana z fragmentem receptora cytokiny gp130 . Kiedy przynęta i zdobycz wchodzą w interakcję, TYK2 fosforyluje miejsca dokowania STAT3 w chimerze zdobyczy, co ostatecznie prowadzi do aktywacji genu reporterowego .
Warianty jedno-, trzy- i jedno-dwu-hybrydowe
Jedna hybryda
Jednohybrydowa odmiana tej techniki została zaprojektowana do badania interakcji białko-DNA i wykorzystuje pojedyncze białko fuzyjne, w którym AD jest bezpośrednio połączona z domeną wiążącą. Jednak domena wiążąca w tym przypadku niekoniecznie ma ustaloną sekwencję, jak w analizie dwuhybrydowej białko-białko, ale może być utworzona przez bibliotekę. Ta biblioteka może być wybrana względem pożądanej sekwencji docelowej, która jest wstawiona w region promotorowy konstruktu genu reporterowego. W systemie selekcji pozytywnej wybiera się zatem domenę wiążącą, która skutecznie wiąże UAS i umożliwia transkrypcję.
Należy zauważyć, że selekcja domen wiążących DNA niekoniecznie jest przeprowadzana przy użyciu systemu jednohybrydowego, ale może być również przeprowadzana przy użyciu systemu dwuhybrydowego, w którym domena wiążąca jest zróżnicowana, a białka przynęty i zdobyczy są utrzymywane na stałym poziomie.
Trzy-hybrydowy
Interakcje RNA-białko badano za pomocą trójhybrydowej odmiany techniki dwuhybrydowej. W tym przypadku hybrydowa cząsteczka RNA służy do łączenia ze sobą dwóch domen fuzyjnych białek – które nie są przeznaczone do interakcji ze sobą, ale raczej z pośrednią cząsteczką RNA (poprzez ich domeny wiążące RNA). Techniki obejmujące białka niefuzyjne, które pełnią podobną funkcję, jak opisano w sekcji „Białka niefuzyjne” powyżej, mogą być również określane jako metody trójhybrydowe.
Jeden-dwa-hybrydy
Jednoczesne stosowanie metod jedno- i dwuhybrydowych (tj. równoczesnej interakcji białko-białko i białko-DNA) jest znane jako podejście jedno-dwuhybrydowe i oczekuje się, że zwiększy rygorystyczność badań przesiewowych.
Organizm gospodarza
Chociaż teoretycznie każda żywa komórka może być wykorzystana jako tło do analizy dwuhybrydowej, istnieją praktyczne względy, które dyktują wybór. Wybrana linia komórkowa powinna być stosunkowo tania i łatwa w hodowli oraz wystarczająco wytrzymała, aby wytrzymać zastosowanie metod badawczych i odczynników. To ostatnie jest szczególnie ważne przy prowadzeniu badań o dużej przepustowości . Dlatego drożdże S. cerevisiae były głównym organizmem gospodarza w badaniach dwuhybrydowych. Jednak nie zawsze jest to idealny system do badania oddziałujących białek z innych organizmów. Komórki drożdży często nie mają takich samych modyfikacji potranslacyjnych, mają inne zastosowanie kodonów lub brakuje im pewnych białek, które są ważne dla prawidłowej ekspresji białek. Aby poradzić sobie z tymi problemami, opracowano kilka nowatorskich systemów dwuhybrydowych. W zależności od zastosowanego systemu do hodowli komórek i umożliwienia selekcji do interakcji stosuje się płytki agarowe lub specyficzną pożywkę wzrostową. Najczęściej stosowaną metodą jest wysiewanie agaru, w którym komórki wysiewa się na pożywkę selekcyjną, aby zobaczyć, jak zachodzi interakcja. Komórki, które nie mają białek oddziałujących, nie powinny przeżyć na tej pożywce selekcyjnej.
S. cerevisiae (drożdże)
Drożdże S. cerevisiae były organizmem modelowym stosowanym podczas tworzenia techniki dwuhybrydowej. Jest powszechnie znany jako system Y2H. Ma kilka cech, które sprawiają, że jest silnym organizmem przyjmującym interakcję, w tym zdolność do tworzenia trzeciorzędowych struktur białkowych, neutralne wewnętrzne pH, zwiększoną zdolność do tworzenia wiązań dwusiarczkowych i glutationu w stanie zredukowanym wśród innych cytozolowych czynników buforowych, aby utrzymać gościnność wewnętrzną środowisko. Model drożdży można manipulować technikami niemolekularnymi, a jego pełna sekwencja genomu jest znana. Systemy drożdży tolerują różne warunki hodowli i ostre chemikalia, których nie można było zastosować w kulturach tkankowych ssaków.
Szereg szczepów drożdży zostało stworzonych specjalnie do badań przesiewowych Y2H, np. Y187 i AH109 , oba produkowane przez Clontech . Wykorzystano również szczepy drożdży R2HMet i BK100.
Candida albicans
C. albicans to drożdże o szczególnej właściwości: tłumaczy kodon CUG raczej na serynę niż na leucynę. Z powodu tego odmiennego wykorzystania kodonów trudno jest zastosować system modelowy S. cerevisiae jako Y2H do sprawdzenia interakcji białko-białko przy użyciugenów C. albicans . Aby zapewnić bardziej natywne środowisko, opracowanodwuhybrydowy (C2H) system C. albicans . Dzięki temu systemowi interakcje białko-białko mogą być badane w samym C. albicans . Ostatnim dodatkiem było stworzenie systemu o wysokiej przepustowości.
E coli
Bakteryjne metody dwuhybrydowe (B2H lub BTH) są zwykle przeprowadzane w E. coli i mają pewną przewagę nad systemami opartymi na drożdżach. Na przykład wyższa wydajność transformacji i szybsze tempo wzrostu sprawia, że E. coli korzysta z większych bibliotek (ponad 108 ). Brak wymagań dotyczących sygnału lokalizacji jądrowej, który ma być zawarty w sekwencji białkowej i zdolność do badania białek, które byłyby toksyczne dla drożdży, mogą również być głównymi czynnikami do rozważenia przy wyborze doświadczalnego organizmu tła.
Aktywność metylacji niektórych białek metylotransferazy DNA E. coli może zakłócać niektóre selekcje białek wiążących DNA. Jeśli można się tego spodziewać, oczywistym rozwiązaniem może być zastosowanie szczepu E. coli , który jest defektywny dla określonej metylotransferazy. B2H może nie być idealny podczas badania eukariotycznych interakcji białko-białko (np. białka ludzkie), ponieważ białka mogą nie fałdować się jak w komórkach eukariotycznych lub mogą nie mieć innego przetwarzania.
Komórki ssaków
W ostatnich latach zaprojektowano system dwuhybrydowy ssaków (M2H) do badania interakcji białko-białko ssaków w środowisku komórkowym, które ściśle naśladuje środowisko białek natywnych. Przejściowo transfekowane komórki ssaków są wykorzystywane w tym systemie do wykrywania interakcji białko-białko. Wykorzystanie linii komórek ssaków do badania interakcji białko-białko ssaków daje przewagę pracy w bardziej natywnym kontekście. Modyfikacje potranslacyjne, fosforylacja, acylacja i glikozylacja są podobne. Wewnątrzkomórkowa lokalizacja białek jest również bardziej poprawna w porównaniu z zastosowaniem drożdżowego systemu dwuhybrydowego. W systemie dwuhybrydowym ssaków możliwe jest również badanie sygnałów wejściowych. Kolejną dużą zaletą jest to, że wyniki można uzyskać w ciągu 48 godzin po transfekcji.
Arabidopsis thaliana
W 2005 roku opracowano system dwóch hybryd w zakładach. Wykorzystując protoplasty interakcji białko-białko A. thaliana można badać u roślin. W ten sposób interakcje można badać w ich rodzimym kontekście. W tym systemie GAL4 AD i BD są pod kontrolą silnego promotora 35S. Interakcja jest mierzona za pomocą reportera GUS. Aby umożliwić przesiewanie o wysokiej przepustowości, wektory zostały dostosowane do bramek. System znany jest jako system protoplast two hybrid (P2H).
Aplysia californica
Zając morski Kalifornica jest organizmem modelowym w neurobiologii do badania m.in. molekularnych mechanizmów pamięci długotrwałej. Aby zbadać interakcje, ważne w neurologii, w bardziej natywnym środowisku opracowano układ dwuhybrydowy w neuronach A californica . W tym systemie stosowane są GAL4 AD i BD.
Bombyks mori
System dwuhybrydowy owadów (I2H) został opracowany w linii komórkowej jedwabnika z larwy lub gąsienicy udomowionej ćmy jedwabnikowej, Bombyx mori (komórki BmN4). Ten system wykorzystuje GAL4 BD i domenę aktywacyjną mysiego NF-κB P65. Oba są pod kontrolą promotora OpIE2.
Aplikacje
Wyznaczanie sekwencji kluczowych dla interakcji
Zmieniając określone aminokwasy przez mutację odpowiednich par zasad DNA w stosowanych plazmidach, można określić znaczenie tych reszt aminokwasowych w utrzymaniu oddziaływania.
Po zastosowaniu metody opartej na komórkach bakteryjnych do selekcji białek wiążących DNA konieczne jest sprawdzenie specyficzności tych domen, ponieważ istnieje granica zakresu, w jakim genom komórki bakteryjnej może pełnić funkcję pochłaniacza dla domen o powinowactwie do innych sekwencje (lub rzeczywiście, ogólne powinowactwo do DNA).
Odkrycie narkotyków i trucizn
Interakcje sygnalizacyjne białko-białko stanowią odpowiednie cele terapeutyczne ze względu na ich specyficzność i wszechobecność. Metoda losowego odkrywania leków wykorzystuje banki związków, które zawierają losowe struktury chemiczne i wymaga wysokowydajnej metody testowania tych struktur w zamierzonym celu.
Komórka wybrana do badania może być specjalnie zaprojektowana tak, aby odzwierciedlała molekularny aspekt, który badacz zamierza zbadać, a następnie wykorzystana do zidentyfikowania nowych środków terapeutycznych dla ludzi lub zwierząt lub środków przeciw szkodnikom.
Oznaczanie funkcji białka
Poprzez określenie partnerów interakcji nieznanych białek można wywnioskować możliwe funkcje tych nowych białek. Można to zrobić przy użyciu pojedynczego znanego białka wobec biblioteki nieznanych białek lub odwrotnie, wybierając z biblioteki znanych białek przy użyciu pojedynczego białka o nieznanej funkcji.
Wybór białka palca cynkowego
Aby wyselekcjonować białka palca cynkowego (ZFP) do inżynierii białek , z powodzeniem zastosowano metody zaadaptowane z dwuhybrydowej techniki przesiewowej. ZFP jest samo w sobie białkiem wiążącym DNA, używanym do budowy niestandardowych domen wiążących DNA, które wiążą się z pożądaną sekwencją DNA.
Stosując gen selekcyjny z pożądaną sekwencją docelową zawartą w UAS i randomizując odpowiednie sekwencje aminokwasowe w celu wytworzenia biblioteki ZFP, można wyselekcjonować komórki, które są gospodarzem oddziaływania DNA-ZFP o wymaganych właściwościach. Każdy ZFP zazwyczaj rozpoznaje tylko 3–4 pary zasad, więc aby zapobiec rozpoznaniu miejsc poza UAS, randomizowany ZFP jest wbudowywany w „rusztowanie” składające się z kolejnych dwóch ZFP o stałej sekwencji. UAS jest zatem zaprojektowany tak, aby zawierał sekwencję docelową stałego rusztowania oprócz sekwencji, dla której wybrano ZFP.
Za pomocą tego systemu można również zbadać szereg innych domen wiążących DNA.
Silne strony
- Ekrany dwuhybrydowe są low-tech; można je przeprowadzić w dowolnym laboratorium bez wyrafinowanego sprzętu.
- Ekrany dwuhybrydowe mogą stanowić ważną pierwszą wskazówkę do identyfikacji partnerów interakcji.
- Test jest skalowalny, co umożliwia przeszukiwanie pod kątem interakcji między wieloma białkami. Co więcej, można ją zautomatyzować, a dzięki zastosowaniu robotów wiele białek może zostać przebadanych pod kątem tysięcy potencjalnie wchodzących w interakcje białek w stosunkowo krótkim czasie. Stosowane są dwa rodzaje dużych ekranów: podejście biblioteczne i podejście macierzowe.
- Dane dwuhybrydowe dla drożdży mogą być podobnej jakości do danych generowanych przez alternatywne podejście oczyszczania współpowinowactwa, a następnie spektrometrii masowej (AP/MS).
Słabości
- Główną krytyką dotyczącą dwuhybrydowego badania przesiewowego interakcji białko-białko drożdży jest możliwość dużej liczby fałszywie dodatnich (i fałszywie ujemnych) identyfikacji. Dokładny odsetek wyników fałszywie dodatnich nie jest znany, ale wcześniejsze szacunki wynosiły aż 70%. To również, częściowo, wyjaśnia często stwierdzane bardzo małe nakładanie się wyników przy użyciu (wysokiej przepustowości) dwuhybrydowego skriningu, zwłaszcza przy użyciu różnych systemów eksperymentalnych.
Powodem tego wysokiego poziomu błędów jest charakterystyka ekranu:
- Niektóre warianty testu wykazują nadekspresję białek fuzyjnych, co może powodować nienaturalne stężenia białek, które prowadzą do nieswoistych (fałszywie dodatnich) wyników.
- Białka hybrydowe są białkami fuzyjnymi; to znaczy, że połączone części mogą hamować pewne interakcje, zwłaszcza jeśli interakcja zachodzi na N-końcu badanego białka (gdzie zazwyczaj przyłączana jest domena wiążąca DNA lub aktywująca).
- Interakcja może nie mieć miejsca w drożdżach, typowym organizmie gospodarza dla Y2H. Na przykład, jeśli białko bakteryjne jest testowane na drożdżach, może brakować mu białka opiekuńczego do prawidłowego fałdowania, które jest obecne tylko w gospodarzu bakteryjnym. Co więcej, białko ssaków czasami nie jest prawidłowo modyfikowane w drożdżach (np. brak fosforylacji ), co może również prowadzić do fałszywych wyników.
- Y2H ma miejsce w jądrze. Jeśli badane białka nie są zlokalizowane w jądrze (ponieważ mają inne sygnały lokalizacyjne), można stwierdzić, że dwa oddziałujące białka nie oddziałują.
- Niektóre białka mogą specyficznie oddziaływać, gdy są współeksprymowane w drożdżach, chociaż w rzeczywistości nigdy nie są obecne w tej samej komórce w tym samym czasie. Jednak w większości przypadków nie można wykluczyć, że takie białka rzeczywiście ulegają ekspresji w pewnych komórkach lub w określonych okolicznościach.
Każdy z tych punktów sam w sobie może prowadzić do fałszywych wyników. Ze względu na połączone efekty wszystkich źródeł błędów drożdże dwuhybrydowe należy interpretować z ostrożnością. Prawdopodobieństwo wygenerowania wyników fałszywie dodatnich oznacza, że wszystkie interakcje należy potwierdzić testem o wysokim stopniu ufności, na przykład koimmunoprecypitacją białek endogennych, co jest trudne w przypadku danych dotyczących interakcji białko-białko na dużą skalę. Alternatywnie dane Y2H można zweryfikować przy użyciu wielu wariantów Y2H lub technik bioinformatycznych. Te ostatnie sprawdzają, czy oddziałujące białka ulegają ekspresji w tym samym czasie, mają pewne wspólne cechy (takie jak adnotacje ontologii genów lub pewne topologie sieci ), mają interakcje homologiczne u innych gatunków.
Zobacz też
- Prezentacja fagowa , alternatywna metoda wykrywania interakcji białko-białko i białko-DNA
- Macierz białkowa , oparta na chipie metoda wykrywania interakcji białko-białko
- Analiza syntetycznych macierzy genetycznych , oparta na drożdżach metoda badania interakcji genów
Bibliografia
Zewnętrzne linki
|
Zasoby biblioteczne o pokazach dwuhybrydowych |
- Szczegóły na temat siostrzanego systemu dwuhybrydowego
- Przegląd drożdżowego systemu dwuhybrydowego opublikowany przez Science Creative Quarterly
- Jednohybrydowe ekrany drożdżowe kompatybilne z bramką
- Animacja wideo Drożdżowego Systemu Dwuhybrydowego
- Two-Hybrid+System+Techniques w Narodowej Bibliotece Medycznej USA Medical Subject Headings (MeSH)
- Drożdże Dwuhybrydowe
- Baza danych BioGrid z interakcjami białko-białko

