Struktura biomolekularna - Biomolecular structure

|

|
Biomolecular konstrukcja jest skomplikowana złożony, trójwymiarowy kształt, który jest utworzony z cząsteczek o białka , DNA lub RNA , i jest istotne dla jego funkcji. Strukturę tych cząsteczek można rozpatrywać w dowolnej z kilku skal długości, od poziomu pojedynczych atomów do relacji między całymi podjednostkami białka . To użyteczne rozróżnienie między skalami jest często wyrażane jako rozkład struktury molekularnej na cztery poziomy: pierwszorzędowy, drugorzędowy, trzeciorzędowy i czwartorzędowy. Rusztowanie dla tej wieloskalowej organizacji cząsteczki powstaje na poziomie wtórnym, gdzie podstawowymi elementami strukturalnymi są różne wiązania wodorowe cząsteczki . Prowadzi to do kilku rozpoznawalnych domen o strukturze białka i struktury kwasu nukleinowego , w tym tych funkcji wtórnej struktury, jak alfa-helisy i arkuszy beta dla białek i szpilki do włosów pętli , wybrzuszenia i pętli wewnętrznej dla kwasów nukleinowych. Terminy struktura pierwszorzędowa , drugorzędowa , trzeciorzędowa i czwartorzędowa zostały wprowadzone przez Kaj Ulrik Linderstrøm-Lang w jego 1951 Lane Medical Lectures na Uniwersytecie Stanforda .
Podstawowa struktura
Podstawowej struktury z biopolimeru to dokładne określenie składu atomowego oraz wiązania chemiczne łączące te atomy (włącznie z stereochemii ). W przypadku typowego nierozgałęzionego, nieusieciowanego biopolimeru (takiego jak cząsteczka typowego białka wewnątrzkomórkowego lub DNA lub RNA ) struktura pierwszorzędowa jest równoważna określeniu sekwencji jego podjednostek monomerycznych , takich jak aminokwasy lub nukleotydy .
Struktura pierwotna jest czasem błędnie nazywana sekwencją pierwotną , ale nie ma takiego terminu, ani równoległego pojęcia sekwencji drugorzędowej lub trzeciorzędowej. Zgodnie z konwencją, struktura pierwszorzędowa białka jest opisywana od końca aminowego (N) do końca karboksylowego (C), podczas gdy struktura pierwszorzędowa cząsteczki DNA lub RNA jest opisywana od końca 5' do końca 3'.
Podstawowej struktury cząsteczki kwasu nukleinowego, odnosi się do dokładnej sekwencji nukleotydów, które obejmują całe cząsteczki. Często struktura pierwotna koduje motywy sekwencji, które mają znaczenie funkcjonalne. Niektóre przykłady takich motywów są: puszki C / D i H / ACA z snoRNAs , LSM miejsce wiązania się w RNA spliceosomalnych jak U1 , U2 , U4 , U5 , U6 , U12 i U3 , w sekwencji Shine-Dalgarno , a z Kozak sekwencja konsensusowa i terminator polimerazy RNA III .
Struktura drugorzędna
Struktura drugorzędowa to wzór wiązań wodorowych w biopolimerze. Określają one ogólną trójwymiarową postać lokalnych segmentów biopolimerów, ale nie opisują globalnej struktury określonych pozycji atomów w przestrzeni trójwymiarowej, które są uważane za strukturę trzeciorzędową . Struktura drugorzędowa jest formalnie zdefiniowana przez wiązania wodorowe biopolimeru, co obserwuje się w strukturze o rozdzielczości atomowej. W białkach strukturę drugorzędową określają wzory wiązań wodorowych między szkieletowymi grupami aminowymi i karboksylowymi (wiązania wodorowe łańcuch boczny-łańcuch główny i łańcuch boczny-łańcuch boczny są nieistotne), gdzie stosowana jest definicja wiązania wodorowego według DSSP . W kwasach nukleinowych strukturę drugorzędową określa wiązanie wodorowe między zasadami azotowymi.
Jednak w przypadku białek wiązanie wodorowe jest skorelowane z innymi cechami strukturalnymi, co doprowadziło do mniej formalnych definicji struktury drugorzędowej. Na przykład helisy mogą przyjmować dwuścienne kąty szkieletu w niektórych regionach wykresu Ramachandrana ; w związku z tym segment reszt o takich kątach dwuściennych jest często nazywany helisą , niezależnie od tego, czy ma prawidłowe wiązania wodorowe. Zaproponowano wiele innych mniej formalnych definicji, często stosujących pojęcia z geometrii różniczkowej krzywych, takie jak krzywizna i skręcanie . Biolodzy strukturalni rozwiązujący nową strukturę o rozdzielczości atomowej czasami przypisują jej drugorzędową strukturę naocznie i zapisują swoje przypisania w odpowiednim pliku Protein Data Bank (PDB).
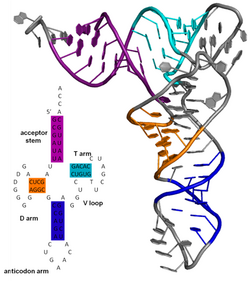
Wtórna struktura cząsteczki kwasu nukleinowego, odnosi się do parowania zasad interakcji wewnątrz jednej cząsteczki lub zestaw cząsteczek oddziałujących. Strukturę drugorzędową biologicznych RNA można często w unikalny sposób rozłożyć na łodygi i pętle. Często te elementy lub ich kombinacje mogą być dalej klasyfikowane, np. tetrapętle , pseudowęzły i pętle łodygi . Istnieje wiele elementów struktury drugorzędowej o znaczeniu funkcjonalnym dla biologicznego RNA. Znane przykłady obejmują pętle łodygi niezależnego od Rho terminatora i transferowy RNA (tRNA) koniczyny. Istnieje niewielki przemysł badaczy próbujących określić drugorzędową strukturę cząsteczek RNA. Podejścia obejmują zarówno metody eksperymentalne, jak i obliczeniowe (patrz także Lista oprogramowania do przewidywania struktury RNA ).
Struktura trzeciorzędowa
Strukturę trzeciorzędową z białka lub inne makrocząsteczki jest jego trójwymiarową strukturę, zgodnie ze współrzędnymi atomowymi. Białka i kwasy nukleinowe fałdują się w złożone trójwymiarowe struktury, które skutkują funkcjami molekuł. Chociaż takie struktury są różnorodne i złożone, często składają się z powtarzających się, rozpoznawalnych motywów i domen struktury trzeciorzędowej, które służą jako molekularne elementy budulcowe. Uważa się, że struktura trzeciorzędowa jest w dużej mierze zdeterminowana przez pierwszorzędową strukturę biomolekuły (jej sekwencja aminokwasów lub nukleotydów ).
Struktura czwartorzędowa
Struktura czwartorzędowa odnosi się do liczby i rozmieszczenia wielu cząsteczek białka w kompleksie wielopodjednostkowym. W przypadku kwasów nukleinowych termin ten jest mniej powszechny, ale może odnosić się do wyższego poziomu organizacji DNA w chromatynie , w tym jego interakcji z histonami , lub do interakcji między oddzielnymi jednostkami RNA w rybosomie lub spliceosomie .
Określanie struktury
Sondowanie struktury to proces, w którym do określania struktury biomolekularnej wykorzystuje się techniki biochemiczne. Ta analiza może być wykorzystana do zdefiniowania wzorców, które można wykorzystać do wnioskowania o strukturze molekularnej, eksperymentalnej analizy struktury i funkcji molekularnej oraz dalszego zrozumienia rozwoju mniejszych cząsteczek do dalszych badań biologicznych. Analizę sondowania struktury można przeprowadzić wieloma różnymi metodami, które obejmują sondowanie chemiczne, sondowanie rodników hydroksylowych, mapowanie interferencji analogów nukleotydów (NAIM) i sondowanie w linii.
Struktury białek i kwasów nukleinowych można określić za pomocą spektroskopii magnetycznego rezonansu jądrowego ( NMR ) lub krystalografii rentgenowskiej lub mikroskopii krioelektronowej pojedynczych cząstek ( crioEM ). Pierwsze opublikowane raporty dotyczące DNA ( Rosalind Franklin i Raymond Gosling w 1953) wzorów dyfrakcji rentgenowskiej A-DNA — a także B-DNA — wykorzystywały analizy oparte na transformacjach funkcji Pattersona, które dostarczały jedynie ograniczonej ilości informacji strukturalnych dla zorientowanych włókna DNA wyizolowane z grasicy cielęcej . Analiza alternatywna została następnie zaproponowana przez Wilkinsa i in. w 1953 dla B-DNA dyfrakcji rentgenowskiej i wzorców rozpraszania uwodnionych, zorientowanych bakteryjnie włókien DNA i główek plemników pstrągów pod względem kwadratów funkcji Bessela . Chociaż forma B-DNA jest najbardziej powszechna w warunkach występujących w komórkach, nie jest to dobrze zdefiniowana konformacja, ale rodzina lub rozmyty zestaw konformacji DNA, które występują przy wysokich poziomach uwodnienia obecnych w wielu różnych żywych komórkach. Ich odpowiednie wzory dyfrakcji i rozpraszania promieniowania rentgenowskiego są charakterystyczne dla parakryształów molekularnych o znacznym stopniu nieuporządkowania (ponad 20%), a struktura nie jest możliwa do zbadania przy użyciu tylko standardowej analizy.
W przeciwieństwie do standardowej analizy, obejmujący tylko transformacji Fouriera z funkcji Bessela i DNA modeli cząsteczkowych , nadal jest stosowane rutynowo do analizy DNA i A-X-ray dyfraktogramy Z-DNA.
Przewidywanie struktury

Przewidywanie struktury biomolekularnej to przewidywanie trójwymiarowej struktury białka na podstawie jego sekwencji aminokwasowej lub kwasu nukleinowego na podstawie jego sekwencji nukleozasad (zasad). Innymi słowy, jest to przewidywanie struktury drugorzędowej i trzeciorzędowej na podstawie jej struktury pierwszorzędowej. Przewidywanie struktury jest odwrotnością biomolekularnych konstrukcji, jak w racjonalnego projektowania , konstrukcji białek , konstrukcji kwasu nukleinowego i inżynierii biomolekularnych .
Przewidywanie struktury białek jest jednym z najważniejszych celów bioinformatyki i chemii teoretycznej . Przewidywanie struktury białka ma duże znaczenie w medycynie (na przykład przy projektowaniu leków ) i biotechnologii (na przykład przy projektowaniu nowych enzymów ). Co dwa lata wydajność obecnych metod jest oceniana w eksperymencie krytycznej oceny przewidywania struktury białek ( CASP ).
Odbyło się również wiele badań bioinformatycznych ukierunkowanych na problem przewidywania struktury RNA. Częstym problemem badaczy pracujących z RNA jest określenie trójwymiarowej struktury cząsteczki na podstawie tylko sekwencji kwasu nukleinowego. Jednak w przypadku RNA znaczna część końcowej struktury jest określona przez strukturę drugorzędową lub wewnątrzcząsteczkowe oddziaływania parowania zasad w cząsteczce. Wskazuje na to wysoka konserwacja par zasad w różnych gatunkach.
Strukturę drugorzędową małych cząsteczek kwasu nukleinowego determinują w dużej mierze silne, lokalne oddziaływania, takie jak wiązania wodorowe i układanie zasad . Sumowanie energii swobodnej dla takich oddziaływań, zwykle metodą najbliższego sąsiedztwa , daje przybliżenie stateczności danej konstrukcji. Najprostszym sposobem znalezienia struktury o najniższej energii swobodnej byłoby wygenerowanie wszystkich możliwych struktur i obliczenie dla nich energii swobodnej, ale liczba możliwych struktur dla sekwencji rośnie wykładniczo wraz z długością cząsteczki. W przypadku dłuższych cząsteczek liczba możliwych struktur drugorzędowych jest ogromna.
Metody kowariancji sekwencji opierają się na istnieniu zestawu danych złożonego z wielu homologicznych sekwencji RNA z pokrewnymi, ale niepodobnymi sekwencjami. Metody te analizują kowariancję poszczególnych miejsc zasad w ewolucji ; utrzymywanie w dwóch szeroko oddzielonych miejscach pary nukleotydów parujących zasady wskazuje na obecność wymaganego strukturalnie wiązania wodorowego między tymi pozycjami. Wykazano, że ogólny problem przewidywania węzłów pseudowęzłowych jest NP-zupełny .
Projekt
Projektowanie biomolekularne można uznać za odwrotność przewidywania struktury. W przewidywaniu struktury struktura jest określana na podstawie znanej sekwencji, podczas gdy przy projektowaniu białka lub kwasu nukleinowego generowana jest sekwencja, która utworzy pożądaną strukturę.
Inne biomolekuły
Inne biocząsteczki, takie jak polisacharydy , polifenole i lipidy , również mogą mieć strukturę wyższego rzędu o konsekwencjach biologicznych.
Zobacz też
- Biomolekularny
- Porównanie oprogramowania do symulacji kwasów nukleinowych
- Struktura genów
- Lista oprogramowania do przewidywania struktury RNA
- Niekodujący RNA