Mały jąderkowy RNA - Small nucleolar RNA
Małe jąderkowe RNA ( snoRNA ) to klasa małych cząsteczek RNA , które przede wszystkim kierują chemicznymi modyfikacjami innych RNA, głównie rybosomalnych RNA , przenoszących RNA i małych jądrowych RNA . Istnieją dwie główne klasy snoRNA, snoRNA C/D box, które są związane z metylacją , oraz snoRNA H/ACA box, które są związane z pseudourydylacją . SnoRNA są powszechnie określane jako kierujące RNA, ale nie należy ich mylić z kierującymi RNA, które kierują edycją RNA w trypanosomach .
Modyfikacje sterowane snoRNA
Po transkrypcji powstające cząsteczki rRNA (nazywane pre-rRNA) przechodzą szereg etapów przetwarzania w celu wygenerowania dojrzałej cząsteczki rRNA. Przed rozszczepieniem przez egzo- i endonukleazy, pre-rRNA podlega złożonemu wzorcowi modyfikacji nukleozydowych. Należą do nich metylacje i pseudourydylacji kierowane przez snoRNA.
- Metylacja to przyłączenie lub podstawienie grupy metylowej do różnych substratów . rRNA człowieka zawiera około 115 modyfikacji grup metylowych. Większość z nich to metylacje 2′O-rybozy (gdzie grupa metylowa jest przyłączona do grupy rybozy).
- Pseudourydyzacja to konwersja ( izomeryzacja ) nukleozydu urydyny do innej formy izomerycznej pseudourydyny (Ψ). Ta modyfikacja polega na obrocie zasady urydyny o 180º wokół jej wiązania glikozylowego do rybozy szkieletu RNA. Po tej rotacji zasada azotowa wnosi atom węgla do wiązania glikozylowego zamiast zwykłego atomu azotu. Korzystnym aspektem tej modyfikacji jest dodatkowy donor wiązania wodorowego dostępny na podstawie. Podczas gdy urydyna tworzy dwa wiązania wodorowe ze swoją parą zasad Watsona-Cricka, adenina, pseudourydyna jest zdolna do tworzenia trzech wiązań wodorowych. Kiedy pseudourydyna jest sparowana zasadowo z adeniną, może również tworzyć inne wiązanie wodorowe, umożliwiając ukształtowanie się złożoności dojrzałej struktury rRNA. Wolny donor wiązania wodorowego często tworzy wiązanie z zasadą, która jest odległa od siebie, tworząc trzeciorzędową strukturę, w której rRNA musi być funkcjonalne. Dojrzałe ludzkie rRNA zawierają około 95 Ψ modyfikacji.
Każda cząsteczka snoRNA działa jako przewodnik tylko dla jednej (lub dwóch) indywidualnych modyfikacji w docelowym RNA. W celu przeprowadzenia modyfikacji, każdy snoRNA łączy się z co najmniej czterema białkami rdzeniowymi w kompleksie RNA/białko określanym jako mała cząstka jąderkowa rybonukleoproteiny (snoRNP). Białka związane z każdym RNA zależą od typu cząsteczki snoRNA (patrz poniżej rodziny przewodników snoRNA). Cząsteczka snoRNA zawiera element antysensowny (odcinek 10–20 nukleotydów ), który jest zasadą komplementarną do sekwencji otaczającej zasadę ( nukleotyd ) ukierunkowaną na modyfikację w cząsteczce pre-RNA. Umożliwia to snoRNP rozpoznawanie docelowego RNA i wiązanie się z nim. Gdy snoRNP zwiąże się z miejscem docelowym, powiązane białka znajdują się we właściwej fizycznej lokalizacji, aby katalizować chemiczną modyfikację docelowej zasady.
Przewodnik po rodzinach snoRNA
Dwa różne typy modyfikacji rRNA (metylacja i pseudourydylacja) są kierowane przez dwie różne rodziny snoRNA. Te rodziny snoRNA są określane jako antysensowne C/D box i H/ACA box snoRNA w oparciu o obecność konserwatywnych motywów sekwencji w snoRNA. Są wyjątki, ale z reguły członkowie box C/D kierują metylacją, a członkowie H/ACA kierują pseudourydylacji. Członkowie każdej rodziny mogą różnić się biogenezą, strukturą i funkcją, ale każda rodzina jest klasyfikowana według następujących ogólnych cech. Aby uzyskać więcej informacji, zobacz recenzję. SnoRNA są klasyfikowane jako małe jądrowe RNA w MeSH . HGNC , we współpracy z snoRNABase i ekspertów w tej dziedzinie, zatwierdziła unikalne nazwy dla ludzkich genów, które kodują snoRNAs.
Skrzynka C/D
SnoRNA C/D box zawierają dwa krótkie motywy konserwatywnej sekwencji, C (RUGAUGA) i D (CUGA), zlokalizowane odpowiednio w pobliżu końców 5' i 3' snoRNA. Krótkie regiony (~ 5 nukleotydów) zlokalizowane powyżej kasety C i poniżej kasety D są zwykle komplementarne do zasad i tworzą strukturę kasety trzpień-box, która zbliża motywy kasety C i D do bliskiej odległości. Wykazano, że ta struktura trzpień-pudełka jest niezbędna do prawidłowej syntezy snoRNA i lokalizacji jąderka. Wiele snoRNA C/D box zawiera również dodatkową, mniej dobrze zakonserwowaną kopię motywów C i D (określanych jako C' i D') zlokalizowaną w centralnej części cząsteczki snoRNA. Konserwatywny region o długości 10-21 nukleotydów powyżej pola D jest komplementarny do miejsca metylacji docelowego RNA i umożliwia snoRNA utworzenie dupleksu RNA z RNA. Modyfikowany nukleotyd w docelowym RNA znajduje się zwykle w piątej pozycji w górę od D box (lub D' box). SnoRNA C/D box wiążą się z czterema konserwatywnymi ewolucyjnie i niezbędnymi białkami — fibrylaryną (Nop1p), NOP56 , NOP58 i SNU13 (białko 15,5 kD u eukariotów; jego archeonowy homolog to L7Ae) — które tworzą rdzeń snoRNP C/D box .
Istnieje eukariotyczny snoRNA C/D box ( snoRNA U3 ), dla którego nie wykazano, aby kierował 2′- O- metylacją. Zamiast tego działa w przetwarzaniu rRNA, kierując cięciem pre-rRNA.
Pudełko H/ACA
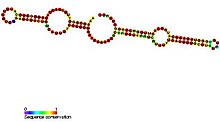
SnoRNA z pudełka H/ACA mają wspólną strukturę drugorzędową składającą się z dwóch szpilek do włosów i dwóch jednoniciowych regionów, określanych jako struktura typu spinka do włosów-zawias-spinka do włosów-ogon. SnoRNA H/ACA zawierają również konserwatywne motywy sekwencji znane jako H box (konsensus ANANNA) i ACA box (ACA). Oba motywy są zwykle zlokalizowane w jednoniciowych obszarach struktury drugorzędowej. Motyw H znajduje się w zawiasie, a motyw ACA w regionie ogona; 3 nukleotydy z końca 3' sekwencji. Regiony spinki do włosów zawierają wewnętrzne wybrzuszenia znane jako pętle rozpoznawania, w których znajdują się antysensowne sekwencje kierujące (zasady komplementarne do sekwencji docelowej). Te sekwencje kierujące zasadniczo wyznaczają położenie urydyny na docelowym rRNA, który ma zostać zmodyfikowany. Ta rozpoznawana sekwencja jest dwuczęściowa (zbudowana z dwóch różnych ramion regionu pętli) i tworzy złożone pseudowęzły z docelowym RNA. SnoRNA kasety H/ACA wiążą się z czterema konserwatywnymi ewolucyjnie i niezbędnymi białkami — dyskeryną (Cbf5p), GAR1 , NHP2 i NOP10 — które tworzą rdzeń snoRNP kasety H/ACA. Dyskeryna jest prawdopodobnie katalitycznym składnikiem kompleksu rybonukleoproteiny (RNP), ponieważ posiada kilka konserwatywnych sekwencji syntazy pseudourydyny i jest blisko spokrewniona z syntazą pseudourydyny, która modyfikuje urydynę w tRNA. W niższych komórkach eukariotycznych, takich jak trypanosomy, podobne RNA występują w postaci pojedynczej struktury spinki do włosów i kasety AGA zamiast kasety ACA na końcu 3' RNA. Podobnie jak Trypanosomy, Entamoeba histolytica ma mieszaną populację snoRNA pudełkowego H/ACA o pojedynczej szpilce i podwójnej szpilce do włosów. Doniesiono, że nastąpiło przetwarzanie snoRNA z podwójnej szpilki do włosów H/ACA w pojedyncze snoRNA o strukturze szpilki do włosów, jednak w przeciwieństwie do trypanosomów, ma on regularny motyw ACA na ogonie 3′. [19]
Składnik RNA ludzkiej telomerazy (hTERC) zawiera domenę H/ACA do tworzenia pre-RNP i jąderkowej lokalizacji samej telomerazy RNP. H/ACA snoRNP został powiązany z rzadką genetyczną chorobą dyskeratozy wrodzonej (DKC) ze względu na jego powiązanie z ludzką telomerazą. Mutacje w składniku białkowym H/ACA snoRNP powodują zmniejszenie fizjologicznych poziomów TERC. Jest to silnie skorelowane z patologią stojącą za DKC, która wydaje się być przede wszystkim chorobą słabego utrzymania telomerów .
Kompozytowe H/ACA i C/D box
Zidentyfikowano niezwykły przewodnik snoRNA U85, który działa zarówno w metylacji 2'-O-rybozy, jak i pseudourydylacji małego jądrowego RNA (snRNA) U5. Ten złożony snoRNA zawiera zarówno domeny kasetowe C/D, jak i H/ACA i wiąże się z białkami specyficznymi dla każdej klasy snoRNA (odpowiednio fibrillarin i Gar1p). Scharakteryzowano teraz więcej złożonych snoRNA.
Stwierdzono, że te złożone snoRNA gromadzą się w podjądrowych organellach nazywanych ciałem Cajala i są określane jako małe RNA specyficzne dla ciała Cajala . Jest to w przeciwieństwie do większości snoRNA C/D box lub H/ACA box, które lokalizują się w jąderku. Sugeruje się, że te specyficzne dla ciała Cajala RNA są zaangażowane w modyfikację spliceosomalnych RNA transkrybowanych przez polimerazę RNA II U1, U2, U4, U5 i U12. Nie wszystkie snoRNA zlokalizowane w ciałach Cajala to złożone snoRNA pudełkowe C/D i H/ACA.
Sieroce snoRNA
Cele dla nowo zidentyfikowanych snoRNA są przewidywane na podstawie komplementarności sekwencji między przypuszczalnymi docelowymi RNA a elementami antysensownymi lub pętlami rozpoznawczymi w sekwencji snoRNA. Jednak istnieje coraz większa liczba „sierocych” przewodników bez żadnych znanych celów RNA, co sugeruje, że w rRNA może być więcej białek lub transkryptów niż wcześniej i/lub że niektóre snoRNA pełnią inne funkcje, niezwiązane z rRNA. Istnieją dowody, że niektóre z tych sierocych snoRNA regulują transkrypty o alternatywnym splicingu. Na przykład wydaje się, że snoRNA C/D box snoRNA SNORD115 reguluje alternatywny splicing mRNA receptora serotoninowego 2C poprzez konserwowany region komplementarności. Przewiduje się , że inny snoRNA C/D box, SNORD116 , który znajduje się w tym samym klastrze co SNORD115, ma 23 możliwe cele w obrębie genów kodujących białka przy użyciu podejścia bioinformatycznego . Spośród nich duża część okazała się podlegać alternatywnemu splicingowi, co sugeruje rolę SNORD116 w regulacji alternatywnego splicingu.
Modyfikacje docelowe
Dokładny wpływ modyfikacji metylacji i pseudourydylacji na funkcję dojrzałych RNA nie jest jeszcze znany. Modyfikacje nie wydają się być niezbędne, ale wiadomo, że subtelnie wzmacniają fałdowanie RNA i interakcję z białkami rybosomalnymi. Na poparcie ich znaczenia modyfikacje miejsc docelowych są zlokalizowane wyłącznie w konserwowanych i funkcjonalnie ważnych domenach dojrzałego RNA i są powszechnie konserwowane wśród odległych eukariontów.
- 2′-O-metylowana ryboza powoduje wzrost konformacji 3′-endo
- Pseudourydyna (psi/Ψ) dodaje kolejną opcję wiązania H.
- Silnie zmetylowany RNA jest chroniony przed hydrolizą. rRNA działa jak rybozym, katalizując własną hydrolizę i splicing.
Organizacja genomowa
SnoRNA są zlokalizowane w genomie w różny sposób. Większość genów snoRNA kręgowców jest zakodowana w intronach genów kodujących białka zaangażowane w syntezę lub translację rybosomów i jest syntetyzowana przez polimerazę II RNA . Wykazano również, że snoRNA są zlokalizowane w regionach międzygenowych, ORF genów kodujących białka i UTR. SnoRNA mogą być również transkrybowane z ich własnych promotorów przez polimerazę RNA II lub III .
Miejsce nadruku
W ludzkim genomie istnieją co najmniej dwa przykłady, w których snoRNA C/D box występują w powtórzeniach tandemowych w obrębie odciskanych loci. Te dwa loci (14q32 na chromosomie 14 i 15q11q13 na chromosomie 15) zostały szeroko scharakteryzowane iw obu regionach znaleziono wiele snoRNA zlokalizowanych w intronach w klastrach blisko spokrewnionych kopii.
W 15q11q13 zidentyfikowano pięć różnych snoRNA ( SNORD64 , SNORD107, SNORD108, SNORD109 (dwie kopie), SNORD116 (29 kopii) i SNORD115 (48 kopii). został zidentyfikowany jako przyczyna zespołu Pradera-Williego, podczas gdy pozyskanie dodatkowych kopii SNORD115 powiązano z autyzmem .
Region 14q32 zawiera powtórzenia dwóch snoRNA SNORD113 (9 kopii) i SNORD114 (31 kopii) w intronach tkankowo-specyficznego transkryptu ncRNA ( MEG8 ). Wykazano, że domena 14q32 ma wspólne cechy genomowe z wdrukowanymi loci 15q11-q13 i sugerowano możliwą rolę powtórzeń tandemowych snoRNA C/D box w ewolucji lub mechanizmie wdrukowanych loci.
Inne funkcje
snoRNA mogą funkcjonować jako miRNA . Wykazano, że ludzki ACA45 jest prawdziwy snoRNA, które mogą być przetworzone na 21- nukleotydów -long dojrzałe miRNA przez endoribonuclease rodziny RNAzy III dicer . Ten produkt snoRNA został wcześniej zidentyfikowany jako mmu-miR-1839 i wykazano, że jest przetwarzany niezależnie od innych droszy endorybonukleazy generującej miRNA . Analizy bioinformatyczne wykazały, że fragmenty przypominające miRNA, które przypuszczalnie pochodzą ze snoRNA, występują w różnych organizmach.
Ostatnio odkryto, że snoRNA mogą mieć funkcje niezwiązane z rRNA. Jednym z takich funkcją jest regulacja alternatywnego składania z trans transkryptu genu, która jest wykonywana przez snoRNA HBII-52 , który jest również znany jako SNORD115.
W listopadzie 2012 r. Schubert i in. ujawnili, że specyficzne RNA kontrolują zagęszczanie i dostępność chromatyny w komórkach Drosophila .
Bibliografia
Zewnętrzne linki
- atlas ludzkiego snoRNA z danych sekwencjonowania małych RNA
- roślinna baza danych snoRNA
- snoRNAbase: baza danych ludzkich H/ACA i C/D box snoRNA
- Baza danych snoRNP
- Baza danych drożdży snoRNA
- wzorzec ekspresji ludzkiego snoRNA
- Strona RFAM dla snoRNA C/D box
- Strona RFAM dla snoRNA H/ACA box
- Strona RFam dla snoRNA scarRNA

